PDF Download - Laborwelt
PDF Download - Laborwelt
PDF Download - Laborwelt
Sie wollen auch ein ePaper? Erhöhen Sie die Reichweite Ihrer Titel.
YUMPU macht aus Druck-PDFs automatisch weboptimierte ePaper, die Google liebt.
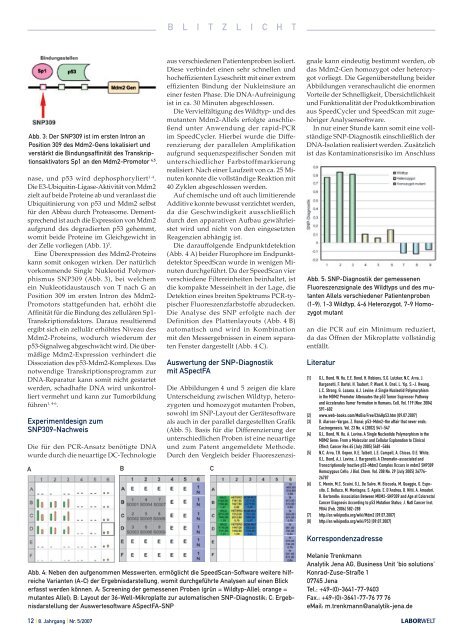
Abb. 3: Der SNP309 ist im ersten Intron an<br />
Position 309 des Mdm2-Gens lokalisiert und<br />
verstärkt die Bindungsaffinität des Transkriptionsaktivators<br />
Sp1 an den Mdm2-Promotor 4,5 .<br />
nase, und p53 wird dephosphoryliert 1-4 .<br />
Die E3-Ubiquitin-Ligase-Aktivität von Mdm2<br />
zielt auf beide Proteine ab und veranlasst die<br />
Ubiquitinierung von p53 und Mdm2 selbst<br />
für den Abbau durch Proteasome. Dementsprechend<br />
ist auch die Expression von Mdm2<br />
aufgrund des degradierten p53 gehemmt,<br />
womit beide Proteine im Gleichgewicht in<br />
der Zelle vorliegen (Abb. 1) 5 .<br />
Eine Überexpression des Mdm2-Proteins<br />
kann somit onkogen wirken. Der natürlich<br />
vorkommende Single Nukleotid Polymorphismus<br />
SNP309 (Abb. 3), bei welchem<br />
ein Nukleotidaustausch von T nach G an<br />
Position 309 im ersten Intron des Mdm2-<br />
Promotors stattgefunden hat, erhöht die<br />
Affinität für die Bindung des zellulären Sp1-<br />
Transkriptionsfaktors. Daraus resultierend<br />
ergibt sich ein zellulär erhöhtes Niveau des<br />
Mdm2-Proteins, wodurch wiederum der<br />
p53-Signalweg abgeschwächt wird. Die übermäßige<br />
Mdm2-Expression verhindert die<br />
Dissoziation des p53-Mdm2-Komplexes. Das<br />
notwendige Transkriptionsprogramm zur<br />
DNA-Reparatur kann somit nicht gestartet<br />
werden, schadhafte DNA wird unkontrolliert<br />
vermehrt und kann zur Tumorbildung<br />
führen 1, 4-6 .<br />
Experimentdesign zum<br />
SNP309-Nachweis<br />
Die für den PCR-Ansatz benötigte DNA<br />
wurde durch die neuartige DC-Technologie<br />
A B C<br />
B L I T Z L I C H T<br />
aus verschiedenen Patientenproben isoliert.<br />
Diese verbindet einen sehr schnellen und<br />
hocheffizienten Lyseschritt mit einer extrem<br />
effizienten Bindung der Nukleinsäure an<br />
einer festen Phase. Die DNA-Aufreinigung<br />
ist in ca. 30 Minuten abgeschlossen.<br />
Die Vervielfältigung des Wildtyp- und des<br />
mutanten Mdm2-Allels erfolgte anschließend<br />
unter Anwendung der rapid-PCR<br />
im SpeedCycler. Hierbei wurde die Differenzierung<br />
der parallelen Amplifikation<br />
aufgrund sequenzspezifischer Sonden mit<br />
unterschiedlicher Farbstoffmarkierung<br />
realisiert. Nach einer Laufzeit von ca. 25 Minuten<br />
konnte die vollständige Reaktion mit<br />
40 Zyklen abgeschlossen werden.<br />
Auf chemische und oft auch limitierende<br />
Additive konnte bewusst verzichtet werden,<br />
da die Geschwindigkeit ausschließlich<br />
durch den apparativen Aufbau gewährleistet<br />
wird und nicht von den eingesetzten<br />
Reagenzien abhängig ist.<br />
Die darauffolgende Endpunktdetektion<br />
(Abb. 4 A) beider Flurophore im Endpunktdetektor<br />
SpeedScan wurde in wenigen Minuten<br />
durchgeführt. Da der SpeedScan vier<br />
verschiedene Filtereinheiten beinhaltet, ist<br />
die kompakte Messeinheit in der Lage, die<br />
Detektion eines breiten Spektrums PCR-typischer<br />
Fluoreszenzfarbstoffe abzudecken.<br />
Die Analyse des SNP erfolgte nach der<br />
Definition des Plattenlayouts (Abb. 4 B)<br />
automatisch und wird in Kombination<br />
mit den Messergebnissen in einem separaten<br />
Fenster dargestellt (Abb. 4 C).<br />
Auswertung der SNP-Diagnostik<br />
mit ASpectFA<br />
Die Abbildungen 4 und 5 zeigen die klare<br />
Unterscheidung zwischen Wildtyp, heterozygoten<br />
und homozygot mutanten Proben,<br />
sowohl im SNP-Layout der Gerätesoftware<br />
als auch in der parallel dargestellten Grafik<br />
(Abb. 5). Basis für die Differenzierung der<br />
unterschiedlichen Proben ist eine neuartige<br />
und zum Patent angemeldete Methode.<br />
Durch den Vergleich beider Fluoreszenzsi-<br />
Abb. 4: Neben den aufgenommen Messwerten, ermöglicht die SpeedScan-Software weitere hilfreiche<br />
Varianten (A-C) der Ergebnisdarstellung, womit durchgeführte Analysen auf einen Blick<br />
erfasst werden können. A: Screening der gemessenen Proben (grün = Wildtyp-Allel; orange =<br />
mutantes Allel); B: Layout der 36-Well-Mikroplatte zur automatischen SNP-Diagnostik; C: Ergebnisdarstellung<br />
der Auswertesoftware ASpectFA-SNP<br />
gnale kann eindeutig bestimmt werden, ob<br />
das Mdm2-Gen homozygot oder heterozygot<br />
vorliegt. Die Gegenüberstellung beider<br />
Abbildungen veranschaulicht die enormen<br />
Vorteile der Schnelligkeit, Übersichtlichkeit<br />
und Funktionalität der Produktkombination<br />
aus SpeedCycler und SpeedScan mit zugehöriger<br />
Analysensoftware.<br />
In nur einer Stunde kann somit eine vollständige<br />
SNP-Diagnostik einschließlich der<br />
DNA-Isolation realisiert werden. Zusätzlich<br />
ist das Kontaminationsrisiko im Anschluss<br />
Abb. 5: SNP-Diagnostik der gemessenen<br />
Fluoreszenzsignale des Wildtyps und des mutanten<br />
Allels verschiedener Patientenproben<br />
(1-9), 1-3 Wildtyp, 4-6 Heterozygot, 7-9 Homozygot<br />
mutant<br />
an die PCR auf ein Minimum reduziert,<br />
da das Öffnen der Mikroplatte vollständig<br />
entfällt.<br />
Literatur<br />
[1] G.L. Bond, W. Hu, E.E. Bond, H. Robions, S.G. Lutzker, N.C. Arva, J.<br />
Bargonetti, F. Bartel, H. Taubert, P. Wuerl, K. Onel, L. Yip, S.-J. Hwang,<br />
L.C. Strong, G. Lozana, A.J. Levine; A Single Nucleotid Polymorphism<br />
in the MDM2 Promotor Attenuates the p53 Tumor Supressor Pathway<br />
and Accelerates Tumor Formation in Humans; Cell, Vol. 119 [Nov. 2004]<br />
591-602<br />
[2] www.web-books.com/MoBio/Free/Ch4Hp53.htm [09.07.2007]<br />
[3] D. Alarcon-Vargas, Z. Ronai; p53-Mdm2-the affair that never ends;<br />
Cacinogenesis, Vol. 23 No. 4 [2002] 541-547<br />
[4] G.L. Bond, W. Hu, A. Levine; A Single Nucleotide Polymorphism in the<br />
MDM2 Gene: From a Molecular and Cellular Explanation to Clinical<br />
Effect; Cancer Res 65 [July 2005] 5481-5484<br />
[5] N.C. Arva, T.R. Gopen, K.E. Talbott, L.E. Campell, A. Chicas, D.E. White,<br />
G.L. Bond, A.J. Levine, J. Bargonetti; A Chromatin-associated and<br />
Transcriptionally Inactive p53-Mdm2 Complex Occurs in mdm2 SNP309<br />
Homozygous Cells; J Biol. Chem. Vol. 208 No. 29 [July 2005] 26776-<br />
26787<br />
[6] C. Menin, M.C. Scaini, G.L. De Salvo, M. Biscuola, M. Quaggio, G. Esposito,<br />
C. Belluco, M. Montagna, S. Agata, E. D‘Andrea, D. Nitti, A. Amadori,<br />
R. Bertorelle; Association Between MDM2-SNP309 and Age at Colorectal<br />
Cancer Diagnosis According to p53 Mutation Status; J. Natl Cancer Inst.<br />
98(4) [Feb. 2006] 582-288<br />
[7] http://en.wikipedia.org/wiki/Mdm2 [09.07.2007]<br />
[8] http://en.wikipedia.org/wiki/P53 [09.07.2007]<br />
Korrespondenzadresse<br />
Melanie Trenkmann<br />
Analytik Jena AG, Business Unit ‘bio solutions’<br />
Konrad-Zuse-Straße 1<br />
07745 Jena<br />
Tel.: +49-(0)-3641-77-9403<br />
Fax.: +49-(0)-3641-77-76 77 76<br />
eMail: m.trenkmann@analytik-jena.de<br />
12 | 8. Jahrgang | Nr. 5/2007 LABORWELT