PDF Download - Laborwelt
PDF Download - Laborwelt
PDF Download - Laborwelt
Erfolgreiche ePaper selbst erstellen
Machen Sie aus Ihren PDF Publikationen ein blätterbares Flipbook mit unserer einzigartigen Google optimierten e-Paper Software.
��������������������������������������������<br />
��������������������<br />
������<br />
��������<br />
���������������������������������������������������<br />
����������������������<br />
����������������������������������������<br />
����������������������������������<br />
��������������������������������<br />
�������������������������������������<br />
���������������������������������<br />
�����������������������������������<br />
�����������������������������������<br />
������������������������������<br />
���������������������������������<br />
���������������������������������<br />
������������������������������������<br />
�������������������������������������<br />
�����������������������������������<br />
�������<br />
������������������������������������<br />
���������<br />
����������������������<br />
����������������������<br />
�������������<br />
�����������������<br />
������������������������<br />
�����������������������<br />
Diagnosepools von 5 ml werden nur 100 µl<br />
Serum oder Plasma pro Tier von maximal 50<br />
Tieren verwendet.<br />
Aus jedem Pool wird ein Aliquot von 200 µl<br />
zur Isolierung viraler RNA verwendet. Hierzu<br />
wird das Aliquot in das im Kit enthaltene<br />
Extraktionsröhrchen übertragen und vorsichtig<br />
mit 200 µl ddH O vermischt. Dieses<br />
2<br />
Extraktionsröhrchen verfügt innen über<br />
eine Beschichtung aus einer vorbereiteten<br />
Mischung fester Lysereagenzien (nicht-chaotroper<br />
Lysepuffer und Proteinase K), Carrier-<br />
Nukleinsäuren und präzise abgemessener<br />
interner RNA/DNA-Extraktionskontrollen.<br />
Die internen Kontrollen wurden mit den<br />
Zielsequenzen zusammen aufgereinigt und<br />
Norm. Fluoro.<br />
ermöglichen die Beurteilung der Extraktionseffizienz<br />
beziehungsweise der PCR-Amplifizierung.<br />
Hierdurch werden die Qualität<br />
der gereinigten Virus-RNA und/oder DNA<br />
sichergestellt und falsch-negative Ergebnisse<br />
0,1 ausgeschlossen.<br />
Nach dem Mischen werden sämtliche<br />
Proben für 15 Minuten bei 65 °C in einen<br />
Schüttelinkubator gegeben, um die Aktivität<br />
0,05 der Proteinase bei der Lyse des Viruskapsids<br />
und der Proteindigestion zu unterstützen.<br />
Zur Entfernung von Proteinresten von den<br />
Virusnukleinsäuren wird darüber hinaus<br />
eine Inkubation von 10 Minuten bei 95 °C<br />
0<br />
empfohlen. Dieser Schritt ist insbesondere<br />
bei sehr stabilen Virusnukleinsäure-Protein-<br />
Komplexen 0 10notwendig. 20 30 40<br />
Während der Lyse ist für jede Probe ein<br />
KingFisher mL-Röhrchenstreifen negative Probe in die<br />
Probenhalterung einzusetzen. Dann werden<br />
die Röhrchenstreifen B bis E gemäß Tabelle 1<br />
mit den mitgelieferten Puffern befüllt. Nach<br />
Abschluss der Lyse werden die Proben in<br />
die KingFisher mL-Röhrchenstreifen über-<br />
tragen (Röhrchen A), und es werden 400 µl<br />
Bindungslösung und 20 µl Magnetpartikellösung<br />
A hinzugefügt und mit jedem Lysat<br />
gemischt, um optimale Bindungsbedingungen<br />
herzustellen.<br />
Unter diesen Bedingungen wird die Virus-RNA<br />
quantitativ an die Magnetpartikel<br />
gebunden. Anschließend werden die Magnetpartikel<br />
in den Waschpuffer R1 und R2<br />
übertragen, um sämtliche Verunreinigungen<br />
wie Proteine, Nukleasen, PCR-Inhibitoren etc.<br />
zu entfernen. Nach der Entfernung des Ethanols<br />
von den Partikeln wird die Virus-RNA<br />
in Elutionspuffer R eluiert und ist bereit zur<br />
weiteren Verwendung.<br />
Nach dem Befüllen der Röhrchenstreifen<br />
Norm. Fluoro.<br />
wird das Tablett im Gerät plaziert und die<br />
Kammspitzen eingesetzt. Die Frontabdekkung<br />
0,05 wird geschlossen und die Proben mit<br />
dem „InviMAG-Virus-KFmL“-Reinigungsprotokoll<br />
verarbeitet. Nach Abschluss des<br />
Programms werden die Eluate zur weiteren<br />
Verwendung gelagert.<br />
positive Probe positive Probe<br />
BVDV-Detektion<br />
Schwellenwert Schwellenwert<br />
Norm. Fluoro.<br />
0,3<br />
0,25<br />
0,2<br />
0,15<br />
0,1<br />
0,05<br />
0<br />
Schwellenwert<br />
Standardmäßig wird ein Aliquot von 5 µl von<br />
jedem Eluat (1/24) zur BVDV-Detektion bei<br />
Probenpools von 50 Tieren verwendet. Primer<br />
und 0Bedingungen<br />
der Echtzeit-One-Step-RT-<br />
PCR beschreiben Gaede et al. Dieses System<br />
ermöglicht eine zuverlässige Detektion selbst<br />
bei nur 0 10 Genomkopien 10 20 pro Reaktion 30 und 40<br />
entspricht den Sensitivitätsanforderungen Zyklus<br />
der Bundesregierung. negative Dies entspricht Probe einer<br />
Verdünnung von mindestens 1 zu 1.000 bei<br />
jeder PI-Probe (Abb. 1).<br />
Des Weiteren wurde ein quantitativer Assay<br />
durchgeführt, um sicherzustellen, dass auch<br />
bei verdünnten Pools eine ausreichende Vi-<br />
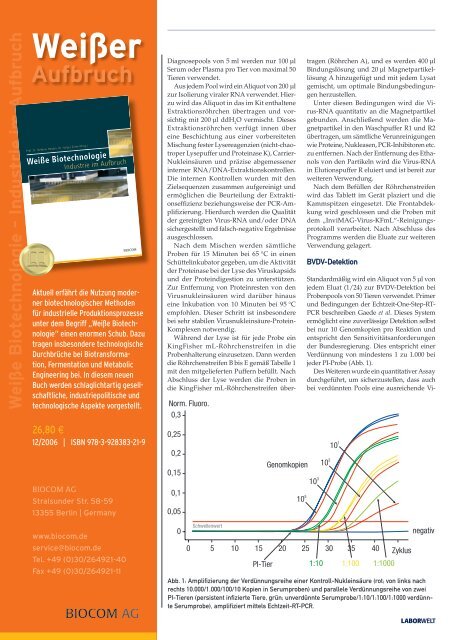
Abb. 1: Amplifizierung der Verdünnungsreihe einer Kontroll-Nukleinsäure (rot; von links nach<br />
rechts 10.000/1.000/100/10 Kopien in Serumproben) und parallele Verdünnungsreihe von zwei<br />
PI-Tieren (persistent infizierte Tiere, grün; unverdünnte Serumprobe/1:10/1:100/1:1000 verdünnte<br />
Serumprobe), amplifiziert mittels Echtzeit-RT-PCR.<br />
22 | 8. Jahrgang | Nr. 5/2007 LABORWELT<br />
Zyklus<br />
Genomkopien<br />
10 5<br />
0 5 10 15 20 25 30 35 40 Zyklus<br />
PI-Tier 1:10 1:100 1:1000<br />
10 3<br />
10 2<br />
10 1<br />
negativ