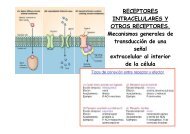
Drosophila - Severo Ochoa - Universidad Autónoma de Madrid
Drosophila - Severo Ochoa - Universidad Autónoma de Madrid
Drosophila - Severo Ochoa - Universidad Autónoma de Madrid
Create successful ePaper yourself
Turn your PDF publications into a flip-book with our unique Google optimized e-Paper software.
Jefe <strong>de</strong> Línea /<br />
Group Lea<strong>de</strong>r:<br />
Mar Ruiz-Gómez<br />
Análisis genético y funcional <strong>de</strong> la<br />
miogénesis en <strong>Drosophila</strong><br />
Genetic and functional analysis of<br />
myogenesis in <strong>Drosophila</strong><br />
A9<br />
Publicaciones<br />
Publications<br />
Postdoctorales /<br />
Postdoctoral:<br />
Marta Carrasco Rando<br />
Becarios Predoctorales /<br />
Predoctoral Fellows:<br />
Silvia Prieto Sánchez<br />
Técnicos <strong>de</strong> Investigación /<br />
Technical Assistance:<br />
Paloma Martín Fernán<strong>de</strong>z<br />
Biología <strong>de</strong>l Desarrollo Developmental Biology<br />
Resumen <strong>de</strong> investigación<br />
Los músculos <strong>de</strong> <strong>Drosophila</strong> y <strong>de</strong> vertebrados están<br />
formados por células sincitiales que se originan por fusión<br />
<strong>de</strong> mioblastos. En <strong>Drosophila</strong> la fusión ocurre entre dos<br />
subtipos <strong>de</strong> mioblastos: los fundadores (MF) y los<br />
competentes en fusión (MCF). Los MFs contienen<br />
información relevante para la adquisición <strong>de</strong> la i<strong>de</strong>ntidad<br />
muscular y para ejecutar el programa <strong>de</strong> diferenciación<br />
terminal, siendo capaces <strong>de</strong> completar miogénesis en<br />
ausencia <strong>de</strong> fusión. Por el contrario, los MCFs en ausencia<br />
<strong>de</strong> fusión no adquieren información acerca <strong>de</strong> su i<strong>de</strong>ntidad<br />
muscular y no completan su programa <strong>de</strong> diferenciación,<br />
muriendo por apoptosis.<br />
En nuestro laboratorio estamos interesados en la<br />
caracterización molecular y funcional <strong>de</strong> los genes<br />
implicados en implementar los programas <strong>de</strong> diferenciación<br />
específicos <strong>de</strong> ambos subtipos <strong>de</strong> mioblastos.<br />
Para ello, utilizamos rastreos genéticos <strong>de</strong> ganancia y<br />
pérdida <strong>de</strong> función para i<strong>de</strong>ntificar posibles candidatos y<br />
una combinación <strong>de</strong> técnicas <strong>de</strong> Biología Molecular y<br />
Celular para su posterior análisis funcional. Recientemente<br />
hemos estudiado la función <strong>de</strong> un nuevo gen, musculus<br />
morbidus (mumo), que codifica una proteína multi-modular<br />
con actividad E3 Ubiquitín ligasa. Su patrón <strong>de</strong> expresión<br />
restringido a MFs sugería un papel importante en la<br />
especificación <strong>de</strong> este subtipo <strong>de</strong> mioblastos. El análisis<br />
fenotípico <strong>de</strong> los alelos <strong>de</strong> falta <strong>de</strong> función generados,<br />
indica que Mumo cumple múltiples funciones durante<br />
miogénesis, como completar la fusión y mantener la<br />
estabilidad <strong>de</strong> las fibras musculares. Así, en ausencia <strong>de</strong><br />
Mumo, los músculos contienen menos núcleos, pier<strong>de</strong>n la<br />
organización sarcomérica y se <strong>de</strong>spren<strong>de</strong>n <strong>de</strong> su sitio <strong>de</strong><br />
anclaje al tendón (ver figura 1). Por lo tanto, mumo confiere<br />
a los MFs una <strong>de</strong> sus propieda<strong>de</strong>s únicas: la capacidad <strong>de</strong><br />
sintetizar sarcómeros estables.<br />
Asimismo, hemos i<strong>de</strong>ntificado otros genes candidatos a<br />
regular distintas etapas <strong>de</strong> la miogénesis.<br />
Research summary<br />
Muscles in <strong>Drosophila</strong> and Vertebrates are syncytial fibres<br />
formed by fusion of myoblasts. In <strong>Drosophila</strong>, fusion takes<br />
place between two distinct populations of myoblasts,<br />
namely foun<strong>de</strong>rs (FM) and fusion competent myoblasts<br />
(FCM). FMs contain the information required for the<br />
acquisition of muscle i<strong>de</strong>ntity and to execute the terminal<br />
differentiation programme. Thus, they are able to complete<br />
myogenesis in the absence of myoblast fusion. On the other<br />
hand, FCMs require fusion to gain information about muscle<br />
i<strong>de</strong>ntity and to accomplish terminal differentiation and they<br />
enter the cell <strong>de</strong>ath programme in the absence of fusion.<br />
Our laboratory aim is to use a combination of genetic,<br />
molecular and cellular techniques to i<strong>de</strong>ntify and characterize<br />
genes involved in the implementation of the differentiation<br />
programmes specific of each myoblast population. Recently,<br />
we have analysed the function of a novel gene musculus<br />
morbidus (mumo). mumo enco<strong>de</strong>s a multi-modular protein<br />
with E3 ubiquitin ligase activity. Its embryonic pattern of<br />
expression exclusive of FMs, suggested that Mumo could<br />
regulate the specification of this sub-type of myoblasts. The<br />
phenotypic analysis of the loss of function alleles generated<br />
indicated that Mumo plays multiple roles during myogenesis,<br />
including regulation of myoblast fusion and maintenance of<br />
sarcomeric stability. Thus, in mumo mutants, muscles contain<br />
fewer nuclei, lack sarcomeric organization and <strong>de</strong>tach from<br />
the apo<strong>de</strong>ma (Figure 1). Therefore, Mumo is required to<br />
confer to FMs one of their unique characteristics, to<br />
synthesize stable sarcomeres.<br />
We have also i<strong>de</strong>ntified other genes that are good candidates<br />
to control distinct steps of the myogenic programme.<br />
Ruiz-Gómez, A., López-Varea, A., Molnar, C., <strong>de</strong> la Calle-Mustienes,<br />
E., Ruiz-Gómez, M., Gómez-Skarmeta, J. L. and <strong>de</strong> Celis, J. F. (2005).<br />
Conserved cross-interactions in <strong>Drosophila</strong> and Xenopus between<br />
Ras/MAPK signaling and the dual-specificity phosphatase MPK3.<br />
Dev. Dyn. 232, 695-708.<br />
Delholm, B., Brown, S., Ray, R.P., Ruiz-Gómez, M., Skaer, H. and<br />
Castelli-Gair Hombría, J. (2005). crossveinless-c is a rhoGAP required<br />
for actin reorganization during morphogenesis.<br />
Development. 132, 2389-2400.<br />
Tesis doctorales<br />
Doctoral Theses<br />
Marta Carrasco Rando (2006). Musculus morbidus una E3 ubiquitín<br />
ligasa <strong>de</strong> <strong>Drosophila</strong>, genera Artrina y mantiene la integridad<br />
<strong>de</strong>l sarcómero.<br />
Figura 1. El gen mumo se requiere para mantener la estabilidad sarcomérica. A, B, La estructura sarcomérica, revelada por la acumulación <strong>de</strong><br />
Ketina-GFP en la banda Z se pier<strong>de</strong> en músculos mutantes mumo (B, comparar con el tipo silvestre mostrado en A). C, D, Patrón muscular <strong>de</strong><br />
embriones silvestres (C) y mutantes mumo (D) revelado por la expresión <strong>de</strong> miosina-GFP. A final <strong>de</strong>l <strong>de</strong>sarrollo embrionario como resultado <strong>de</strong> la<br />
inestabilidad sarcomérica, la mayoría <strong>de</strong> los músculos se han <strong>de</strong>sprendido <strong>de</strong>l tendón. E, Representación esquemática <strong>de</strong>l patrón muscular<br />
abdominal. El código <strong>de</strong> colores indica los músculos que se <strong>de</strong>spren<strong>de</strong>n con mayor frecuencia (blanco, 100%, rojo, menos <strong>de</strong> 10% <strong>de</strong> los casos).<br />
CBM 2005/2006<br />
34<br />
Figure 1. <strong>Drosophila</strong> mumo is required for sarcomeric stability. A, B, Sarcomeric structure revealed by the accumulation of Kettin-GFP in the Z<br />
band is lost in mumo mutant muscles (B, compare to wild type in A). C, D, Muscle pattern of wild type (C) and mumo mutant (D) embryos<br />
revealed by the expression of myosin-GFP. As a consequence of sarcomeric instability most of the muscles <strong>de</strong>tach from the apo<strong>de</strong>ma at the<br />
end of embryogenesis. E, The scheme represents the abdominal muscle pattern, and indicates by a colour co<strong>de</strong> the <strong>de</strong>tachment phenotype<br />
(White indicates 100% <strong>de</strong>tachment, red, less than 10% <strong>de</strong>tachment).<br />
35