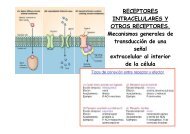
Drosophila - Severo Ochoa - Universidad Autónoma de Madrid
Drosophila - Severo Ochoa - Universidad Autónoma de Madrid
Drosophila - Severo Ochoa - Universidad Autónoma de Madrid
You also want an ePaper? Increase the reach of your titles
YUMPU automatically turns print PDFs into web optimized ePapers that Google loves.
Jefe <strong>de</strong> Línea /<br />
Group Lea<strong>de</strong>r:<br />
José Berenguer<br />
Postdoctorales /<br />
Postdoctoral:<br />
Daniel Vega Mendoza<br />
Becarios Predoctorales /<br />
Predoctoral Fellows:<br />
Felipe Cava<br />
Emilio Blas<br />
Eloy Ferreras<br />
Fe<strong>de</strong>rico Acosta<br />
Zahra Chahlafi<br />
Científicos visitantes /<br />
Visiting Scientist:<br />
Nuno Borges<br />
Elisa Trivelloni<br />
Die<strong>de</strong>rik Johannes Opperman<br />
Regulación <strong>de</strong> la expresión génica Regulation of gene expression<br />
Biotecnología y genética<br />
<strong>de</strong> bacterias termófilas extremas<br />
Resumen <strong>de</strong> investigación<br />
En nuestro grupo empleamos bacterias termófilas extremas<br />
<strong>de</strong>l género Thermus como mo<strong>de</strong>lo <strong>de</strong> laboratorio, y<br />
tratamos <strong>de</strong> <strong>de</strong>scifrar aspectos <strong>de</strong> su biología, y <strong>de</strong><br />
<strong>de</strong>sarrollar en paralelo herramientas genéticas y métodos<br />
que permitan su manipulación y empleo como sistemas <strong>de</strong><br />
producción <strong>de</strong> enzimas o <strong>de</strong> selección <strong>de</strong> formas<br />
termoestables <strong>de</strong> éstas.<br />
Nuestro trabajo en los últimos dos años se ha centrado en<br />
el estudio <strong>de</strong> la regulación y maduración <strong>de</strong> las enzimas<br />
respiratorias codificadas por un elemento genético<br />
transferible (NCE) que confiere al portador la capacidad <strong>de</strong><br />
crecer anaeróbicamente con nitrato como aceptor <strong>de</strong><br />
electrones. Hemos <strong>de</strong>mostrado que la nitrato reductasa<br />
(Nar) codificada por el NCE posee un citocromo c, ausente<br />
en sus homólogas mesófilas, que es necesario para su<br />
maduración y unión a la membrana. Estos datos sugieren<br />
que esta enzima podría constituir el ancestro común a<br />
nitrato reductasas <strong>de</strong> membrana y periplásmicas presentes<br />
en bacterias mo<strong>de</strong>rnas. A nivel <strong>de</strong> regulación, hemos<br />
encontrado que el NCE codifica dos homólogos <strong>de</strong> factores<br />
<strong>de</strong> transcripción <strong>de</strong> las familias CRP y AfsR necesarios para<br />
la expresión <strong>de</strong>l sistema <strong>de</strong> respiración <strong>de</strong> nitrato en<br />
respuesta a la presencia <strong>de</strong> éste compuesto y a la ausencia<br />
simultánea <strong>de</strong> O2. En los dos próximos años preten<strong>de</strong>mos<br />
<strong>de</strong>scifrar la relación entre estos reguladores y los<br />
mecanismos <strong>de</strong> respuesta a estas señales.<br />
A un nivel más aplicado, hemos <strong>de</strong>sarrollado vectores para<br />
expresar enzimas en T. thermophilus <strong>de</strong> una forma<br />
competitiva frente a los sistemas <strong>de</strong> expresión empleados<br />
en E. coli, y vectores <strong>de</strong> plegamiento a alta temperatura con<br />
los que preten<strong>de</strong>mos estabilizar enzimas y proteínas<br />
termosensibles. Estas nuevas herramientas nos permiten<br />
plantearnos proyectos <strong>de</strong> investigación en colaboración<br />
con grupos <strong>de</strong> investigación y con empresas<br />
biotecnológicas. Como ejemplo, hemos contribuido a<br />
i<strong>de</strong>ntificar la función biológica <strong>de</strong>l factor <strong>de</strong> elongación <strong>de</strong> la<br />
transcripción GfhI <strong>de</strong> T. thermophilus (Fig. 1).<br />
Biothechnology and genetic<br />
of extreme thermophilic bacteria<br />
Research summary<br />
We use extreme thermophilic bacteria of the genus Thermus<br />
to uncover specific aspects of its biology and to <strong>de</strong>velop<br />
methods to allow its genetic analysis and use as “cell<br />
factories”, both for the production of thermozimes and for the<br />
selection of thermostable forms from thermosensitive ones.<br />
During the last two years we have focused our attention on<br />
the study of the regulation and maturation of the respiratory<br />
enzymes enco<strong>de</strong>d by a conjugative element (NCE) that<br />
confers the capability to grow anaerobically with nitrate as<br />
electron acceptor to its host. We have <strong>de</strong>monstrated that the<br />
nitrate reductase (Nar) enco<strong>de</strong>d by this NCE contains a<br />
cytochrome c, absent from their mesophilic homologues,<br />
that is required for the binding to the membrane and<br />
maturation of the Nar. These results suggest that Nar could<br />
constitute an ancestor of membrane and periplasmic nitrate<br />
reductases present in mo<strong>de</strong>rn bacteria. At the regulatory<br />
level, we have shown that the NCE enco<strong>de</strong>s two<br />
homologues to transcription factors of the CRP and AfsR<br />
families that are required for the expression of the anerobic<br />
respiratory system in response to nitrate and to the absence<br />
of O2. In the next two years we plan to <strong>de</strong>cipher the<br />
relationship between these regulators and the mechanisms<br />
responsive to these two signals.<br />
At a more applied level, we have <strong>de</strong>veloped vectors that<br />
allow us to express enzymes in T. thermophilus in a<br />
competitive way compared to the expression in E. coli. We<br />
have <strong>de</strong>veloped also a high temperature folding vector with<br />
which we plan to stabilize thermo-sensitive enzymes and<br />
proteins. These new tools allow us to consi<strong>de</strong>r research<br />
projects in collaboration with other groups and with biotech<br />
companies. As an example, we have contributed to<br />
elucidate the biological function of the transcription<br />
elongation factor GfhI from T. thermophilus (Fig. 1).<br />
E5<br />
Publicaciones<br />
Publications<br />
Moreno, R., Haro, A., Castellanos, A. and Berenguer, J. (2005). Highlevel<br />
overproduction of His-tagged Tth DNA polymerase in Thermus<br />
thermophilus. Appl Environ Microbiol. 71: 591-593.<br />
Zafra, O., Cava, F., Blasco, F., Magalon, A. and Berenguer, J. (2005).<br />
Membrane-associated maturation of the heterotetrameric nitrate<br />
reductase of Thermus thermophilus. J. Bacteriol. 187: 3990-3996.<br />
Cava, F. and Berenguer, J. (2006). Biochemical and regulatory<br />
properties of a respiratory island enco<strong>de</strong>d by a conjugative plasmid in<br />
the extreme thermophile Thermus thermophilus.<br />
Biochem Soc Trans. 34: 97-100.<br />
Laptenko, O., Kim, S.S., Lee, J., Starodubtseva, M,, Cava, F.,<br />
Berenguer, J., Kong, X.P. and Borukhov, S. (2006). pH-<strong>de</strong>pen<strong>de</strong>nt<br />
conformational switch activates the inhibitor of transcription<br />
elongation. EMBO J. 25: 2131-2141.<br />
Patentes<br />
Patents<br />
Nuevo marcador <strong>de</strong> selección termoestable para la manipulación<br />
genética <strong>de</strong> Thermus spp. Depósito Nº 200603279. España.<br />
Colaboraciones con la industria<br />
Doctoral Theses<br />
Biotools B & M (<strong>Madrid</strong>) “Efecto <strong>de</strong> proteínas <strong>de</strong> Thermus<br />
thermophilus recombinantes y nativas en reacciones <strong>de</strong> PCR<br />
cualitativas y cuantitativas. Mo<strong>de</strong>lización en la <strong>de</strong>tección <strong>de</strong><br />
Staphylococcus aureus y sus forma resistentes a meticilina (MRSA)”<br />
Biometho<strong>de</strong>s (Evry, Francia) Project THR: A new technology<br />
to improve the thermostability of proteins.<br />
Figura 1. Efecto <strong>de</strong> la <strong>de</strong>leción <strong>de</strong> los factores <strong>de</strong> elongación <strong>de</strong> la transcripción GreA (activador) y Gfh1 (inhibidor) en la morfología <strong>de</strong><br />
Thermus thermophilus. Se muestra la ausencia <strong>de</strong> las proteínas GreA y Gfh1 en mutantes sencillos y dobles obtenidos mediante<br />
el uso <strong>de</strong> un vector <strong>de</strong> inserción-escisión, así como el efecto <strong>de</strong> tales mutaciones en la morfología <strong>de</strong> cada mutante (rojo) comparado<br />
con la estirpe silvestre (ver<strong>de</strong>). Se pue<strong>de</strong> apreciar el pequeño tamaño <strong>de</strong> los mutantes ∆greA, la morfología aberrante <strong>de</strong> los mutantes ∆gfh1<br />
y la formación <strong>de</strong> agregados multicelulares por el doble mutante.<br />
CBM 2005/2006<br />
122<br />
Figure 1. Effect of the <strong>de</strong>letion of the transcription elongation factors GreA (activator) and Gfh1 (inhibitor) on the morphology of Thermus<br />
thermophilus. Figure shows the absence of the GreA and Gfh1 proteins in single and double mutants obtained through the use of an insertionexcision<br />
vector, and also the effects of such mutations on the cell morphology of each mutant (red) compared to the wild type (green).<br />
It can be observed the small size of the ∆greA mutant, the aberrant morphology of the ∆gfh1 mutant and the formation<br />
of huge cell aggregates in the double mutant.<br />
123