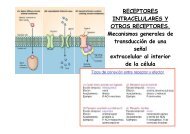
Drosophila - Severo Ochoa - Universidad Autónoma de Madrid
Drosophila - Severo Ochoa - Universidad Autónoma de Madrid
Drosophila - Severo Ochoa - Universidad Autónoma de Madrid
Create successful ePaper yourself
Turn your PDF publications into a flip-book with our unique Google optimized e-Paper software.
Jefes <strong>de</strong> Línea /<br />
Group Lea<strong>de</strong>rs:<br />
Juan Pedro García Ballesta<br />
Miguel Remacha Moreno<br />
Estructura y función <strong>de</strong>l ribosoma<br />
Ribosome structure and function<br />
E10<br />
Publicaciones<br />
Personal Científico /<br />
Scientific Staff:<br />
Antonio Jiménez Díaz<br />
Cruz Santos Tejedor<br />
Francisco Martínez Azorín<br />
Becarios Predoctorales /<br />
Predoctoral Fellows:<br />
Alberto García Marcos<br />
Verónica Briceño Domínguez<br />
Jesús Revuelta Cervantes<br />
María Rodríguez Mateos<br />
Rosario Francisco Velilla<br />
David Cár<strong>de</strong>nas Sanjur<br />
Estudiantes /<br />
Un<strong>de</strong>rgraduated Stu<strong>de</strong>nts:<br />
Belén Rebollo<br />
Vanesa Arribas<br />
Patricia Cerón<br />
Científicos Visitantes /<br />
Visiting Scientists:<br />
E. Gratton<br />
(LFD, <strong>Universidad</strong> <strong>de</strong> Illinois,<br />
Urbana, USA.)<br />
S. Kouyanou<br />
(Departamento <strong>de</strong> Biología,<br />
<strong>Universidad</strong> <strong>de</strong> Atenas, Grecia.)<br />
Sobona Sharma<br />
(Tata Institut, Bombay, India.)<br />
Wanxiang Xu<br />
(Fudan University,<br />
Shanghai, R.P. China.)<br />
Ming F. Tam<br />
(Institute of Molecular Biology,<br />
Aca<strong>de</strong>mia Sinica, Taiwan.)<br />
J. Woolford<br />
(Department of Biological<br />
Sciences, Carnegie Mellon<br />
University, Pittsburg, USA.)<br />
Nilgun Tumer<br />
(Biotechnology Center, Rutgers<br />
University, New Jersey, USA.)<br />
Asistencia Técnica /<br />
Tecnical Assistance:<br />
Mari Carmen Fernán<strong>de</strong>z Moyano<br />
Regulación <strong>de</strong> la expresión génica Regulation of gene expression<br />
Resumen <strong>de</strong> investigación<br />
El ribosoma como regulador <strong>de</strong> la traducción en eucariontes:<br />
Papel <strong>de</strong>l tallo ribosómico.<br />
En eucariontes, el ribosoma no es solamente el elemento<br />
activo central <strong>de</strong>l mecanismo <strong>de</strong> traducción sino que tiene<br />
funciones reguladoras <strong>de</strong> la síntesis proteica. Nuestros<br />
resultados previos han indicado que el tallo ribosómico,<br />
esencial para la actividad <strong>de</strong> los factores solubles, también<br />
pue<strong>de</strong> controlar la traducción <strong>de</strong> <strong>de</strong>terminados mRNAs. A<br />
pesar <strong>de</strong> su importancia este dominio ribosómico es poco<br />
conocido a nivel molecular y por ello intentamos aclarar<br />
aspectos estructurales y funcionales que permitan<br />
compren<strong>de</strong>r especialmente su papel regulador, usando<br />
como mo<strong>de</strong>lo S. cerevisiae y cultivos celulares humanos.<br />
Estructura: Se están estudiando las interacciones<br />
extraordinariamente dinámicas entre las cinco proteínas,<br />
P0, P1α, P1β, P2α y P2β, que forman el tallo <strong>de</strong> la levadura,<br />
y con la proteína L12, asociada a la base <strong>de</strong>l mismo.<br />
Mediante “crosslinking”, cromatografía <strong>de</strong> afinidad con<br />
proteínas etiquetadas, la técnica <strong>de</strong>l doble híbrido, y la<br />
expresión <strong>de</strong> proteínas manipuladas genéticamente, se<br />
están caracterizando los sitios <strong>de</strong> interacción en las<br />
diferentes proteínas y se está analizando su asociación con<br />
otros componentes <strong>de</strong> la maquinaria <strong>de</strong> traducción.<br />
Funcion: Se están i<strong>de</strong>ntificando mRNAs cuya traducción<br />
está afectadas por modificaciones en el tallo ribosómico,<br />
para investigar sobre ellos el mecanismo <strong>de</strong> regulación a<br />
nivel molecular. En S. cerevisiae usamos mutantes con los<br />
genes <strong>de</strong>l tallo ribosómico eliminados, y en cultivos<br />
celulares humanos se inhibe la expresión <strong>de</strong> los mismos<br />
genes usando iRNAs.<br />
Ensamblaje: El ensamblaje <strong>de</strong>l tallo tiene un papel<br />
importante en su función reguladora. La proteína P0 se une<br />
a las partículas preribosómicas en el nucleolo en un proceso<br />
<strong>de</strong> alguna forma controlado por la proteína Mrt4. Las<br />
proteínas P1 y P2 se asocian reversiblemente al ribosoma en<br />
el citoplasma. Se están i<strong>de</strong>ntificando los factores implicados<br />
en este proceso caracterizando partículas pre-ribosómicas<br />
aisladas mediante la técnica <strong>de</strong>l TAP.<br />
Antibióticos: Se está investigando la interacción <strong>de</strong>l<br />
tallo ribosómico con antibióticos que lo inhiben<br />
(sordarinas, thiostrepton).<br />
Research summary<br />
The ribosome as translation regulator in eukaryotes: Role of<br />
the ribosomal stalk.<br />
In eukaryotes, the ribosome is the central active component<br />
of the translation mechanism and also an important<br />
regulatory element of the protein biosynthesis. Our previous<br />
results have shown that the ribosomal stalk, which is<br />
essential for soluble factors activity, can also control the<br />
translation of some mRNAs. In spite of its functional<br />
relevance this ribosomal domain is poorly un<strong>de</strong>rstood at the<br />
molecular level. We are trying to clarify functional and<br />
structural aspects that can help to un<strong>de</strong>rstand the ribosomal<br />
stalk regulatory role using as a mo<strong>de</strong>l S. cerevisiae and<br />
human cell cultures.<br />
Structure: We are studying the extraordinarily dynamic<br />
interactions existing among the five proteins, P0, P1α, P1β,<br />
P2α y P2β, that form the yeast stalk and with protein L12,<br />
which is associated to the stalk base. Using cross-linking,<br />
affinity chromatography, the two-hybrid system and<br />
genetically manipulated proteins, the interaction sites in the<br />
different stalk proteins are being characterized. Similarly, the<br />
association of these proteins with other translation<br />
machinery components is being analyzed.<br />
Function: We are trying to i<strong>de</strong>ntify mRNAs whose translation<br />
is affected by changes in the ribosomal stalk in or<strong>de</strong>r to<br />
investigate in <strong>de</strong>tail the regulatory mechanism at the<br />
molecular level. In S. cerevisiae we are using stalk gene<br />
<strong>de</strong>letion mutants while in human cell culture the expression<br />
of the corresponding genes is suppressed by iRNA.<br />
Assembly: The assembly process has an important role in<br />
the ribosomal stalk regulatory function. Protein P0<br />
associates to the pre-ribosomal particles in the nucleolus in<br />
a process somehow controlled by the assembly protein<br />
Mrt4. In contrast, proteins P1 and P2 reversibly bind to the<br />
ribosome in the cytoplasm. We are trying to i<strong>de</strong>ntify what<br />
factors are involved in this process characterizing preribosomal<br />
particles purified by TAP.<br />
Antibiotics: The mo<strong>de</strong> of action of ribosomal stalk inhibitors<br />
(sordarins, thiostrepton) is also being studied.<br />
Publications<br />
Perez-Fernan<strong>de</strong>z, J., Remacha, M. and Ballesta, J.P.G. (2005). The<br />
acidic protein binding site is partially hid<strong>de</strong>n in the free S. cerevisiae<br />
ribosomal stalk protein P0. Biochemistry. 44, 5532-5540.<br />
Aruna, K., Chakraborti, T., Rao, P., Santos, C., Ballesta, J.P.G. and<br />
Sharma, S. (2005). Functional complementation of yeast ribosomal P0<br />
protein with Plasmodium falciparum P0. Gene. 357, 9-17<br />
Santos, C. and Ballesta, J.P.G. (2005). Characterization of the 26S<br />
rRNA binding domain in Saccharomyces cerevisiae ribosomal stalk<br />
phosphoprotein P0. Mol Microbiol. 58, 217-226.<br />
De la Cruz, J., Sanz-Martínez, E. and Remacha, M. (2005).<br />
The essential WD-repeat protein Rsa4p is required for rRNA<br />
processing and intra-nuclear transport of 60S ribosomal subunits.<br />
Nucleic Acids Res. 33, 5728-5739.<br />
Qiu, D-Y., Parada, P., García Marcos, A., Cár<strong>de</strong>nas, D., Remacha, M.<br />
and Ballesta, J.P.G. (2006). Different roles of P1 and P2 S. cerevisiae<br />
ribosomal stalk proteins revealed by cross-linking.<br />
Mol. Microbiol. 62, 1191.<br />
Tesis doctorales<br />
Doctoral Theses<br />
Alberto García Marcos. (2005). Estudios in vitro e in vivo <strong>de</strong>l tallo<br />
ribosómico <strong>de</strong> S. cerevisiae mediante técnicas biofísicas y bioquímicas.<br />
<strong>Universidad</strong> Autónoma <strong>de</strong> <strong>Madrid</strong>. Sobresaliente cum lau<strong>de</strong>.<br />
Figura 1. Complejo <strong>de</strong> la proteína <strong>de</strong>l tallo ribosómico L11 <strong>de</strong> E. coli (ver<strong>de</strong>), el rRNA <strong>de</strong>l sitio asociado a la GTPasa (GAR) en el 26srRNA <strong>de</strong> S. cerevisiae (amarillo)<br />
y el antibiótico thiostrepton (blanco). Se marcan los residuos <strong>de</strong> Prolina (rojo) <strong>de</strong>l dominio carboxilo terminal (CTD) <strong>de</strong> L11 y los nucleótidos A1095 (azul claro) y G1067<br />
(magenta) directamente relacionados con la unión <strong>de</strong>l antibiótico. También se marcan los aminoácidos Gly-130 y Thr-131 (azul oscuro) y los nucleótidos U-1060 y A1088<br />
(naranja) que juegan un papel clave en la asociación <strong>de</strong> la proteína con el rRNA. Los aminoácidos y nucleótidos están numerados <strong>de</strong> acuerdo con las secuencias <strong>de</strong><br />
E.coli. La estructura se ha mo<strong>de</strong>lado en colaboración con la Unidad <strong>de</strong> Bioinformática <strong>de</strong>l CBMSO usando mo<strong>de</strong>los cristalinos bacterianos disponibles.<br />
CBM 2005/2006<br />
132<br />
Figure 1.Complex of E.coli stalk ribosomal protein L11 (green), the S. cerevisiae GTPase associated region (GAR) of the 26srRNA (yellow) and thiostrepton (white).<br />
The L11 CTD Pro residues (red) and nucleoti<strong>de</strong>s A1095 (cyan) and G1067 (magenta) directly involved in the antibiotic binding are marked. Similarly marked are residues<br />
Gly-130 and Thr-131 (blue) and the nucleoti<strong>de</strong> pair U1060-A1088 (orange), which are highly important for the association of L11 with the GAR region.<br />
The E.coli amino acids and nucleoti<strong>de</strong> numbering is used. The structure has been mo<strong>de</strong>lled in collaboration with the CBMSO Bioinformatics Service.<br />
133