Apri file allegato (risultatiricerca.pdf) - Cra
Apri file allegato (risultatiricerca.pdf) - Cra
Apri file allegato (risultatiricerca.pdf) - Cra
Create successful ePaper yourself
Turn your PDF publications into a flip-book with our unique Google optimized e-Paper software.
CONCLUSIONI<br />
Per una migliore identificazione e tracciabilità dei lieviti appartenenti al genere Saccharomyces, è stato<br />
messo a punto un nuovo approccio enzimatico-molecolare basato sull’analisi delle alcool deidrogenasi<br />
(ADH), un sistema genico che risulta essere altamente conservato nei lieviti nel corso dell’evoluzione.<br />
Ai fini dello studio della variabilità tra le varie specie, il sistema ADH presenta il doppio vantaggio di poter<br />
esser analizzato sia a livello del DNA dei geni che lo costituiscono che dei loro prodotti enzimatici.<br />
Infatti, da quanto scaturisce dalla nostra indagine, utilizzando primers basati sulle sequenze note dei<br />
geni ADH del ceppo di S. cerevisiae di controllo si possono ottenere frammenti di DNA di taglia diversa<br />
specifici per ciascun gene. Allo scopo di ottenere più velocemente un’impronta molecolare dei ceppi<br />
in esame, è possibile effettuare l’amplificazione di tutti e quattro i geni contemporaneamente mediante<br />
PCR multiplex.<br />
Inoltre, la successiva digestione dei prodotti di PCR con l’enzima Sau3AI consente di ottenere un<br />
polimorfismo di restrizione più dettagliato per ciascun lievito da analizzare.<br />
Una volta rilevata la presenza/assenza dei singoli geni ADH, è possibile procedere alla conferma dei<br />
risultati ottenuti sulla base dell’analisi del DNA mediante confronto con quelli ottenuti dallo studio delle<br />
attività enzimatiche ADH codificate dai singoli geni.<br />
Infatti, l’analisi degli zimogrammi ADH ottenuti nell’ambito del presente progetto per ciascun ceppo ha<br />
permesso di confermare che:<br />
a) l’assenza dell’amplificato di uno specifico gene è sempre associata alla mancanza del rispettivo<br />
enzima, a conferma che i primers utilizzati sono altamente specifici per ciascun gene.<br />
b) la presenza/assenza degli enzimi ADH in un determinato ceppo ben si correla con il polimorfismo<br />
di restrizione del relativo DNA.<br />
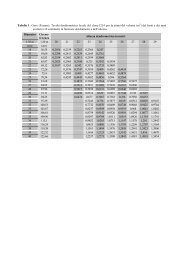
Riassumendo i risultati, l’analisi combinata DNA-enzima suggerisce che i ceppi di chevalieri e italicus<br />
della collezione di Perugia mostrano la presenza di tutti i geni ADH e, pertanto, possono essere<br />
considerati delle varietà di cerevisiae. Al contrario, i ceppi B1 e B2 (S.bayanus) e U2 (S.uvarum)<br />
della stessa collezione risultano privi del gene ADH3. Inoltre, questi tre ceppi presentano un profilo<br />
RFLP e uno zimogramma tra loro sovrapponibili ma molto diversi da quelli delle altre specie di lieviti<br />
analizzati. Questi dati suggeriscono che B1, B2 e U2, classificati sulla base delle loro caratteristiche<br />
fisio-morfologiche in specie diverse del genere Saccharomyces, possono invece appartenere alla stessa<br />
variante di specie.<br />
E’ interessante inoltre sottolineare che il pattern enzimatico del ceppo A-270 di uvarum della collezione<br />
di Velletri, privo del gene ADH3, è sovrapponibile con quello dei ceppi B1, B2 e U2 della collezione di<br />
Perugia.<br />
Al contrario, il ceppo C-S6u uvarum della collezione di Velletri mostra uno zimogramma che somiglia<br />
in parte a quello al ceppo A-270 uvarum e in parte a quello dei cerevisiae var. bayanus della stessa<br />
collezione.<br />
Questo dato conferma la capacità discriminante del nostro metodo dal momento che C-S6u, classificato<br />
1