in Scientia Halensis
in Scientia Halensis
in Scientia Halensis
Sie wollen auch ein ePaper? Erhöhen Sie die Reichweite Ihrer Titel.
YUMPU macht aus Druck-PDFs automatisch weboptimierte ePaper, die Google liebt.
scientia halensis 4/2002<br />
....................................................................................<br />
Fachbereich Biologie<br />
................................................................................<br />
verb<strong>in</strong>dungen, die Synthese Pathogen-<strong>in</strong>duzierter<br />
Prote<strong>in</strong>e, die Verdickung und Quer-<br />
38<br />
vernetzung der Zellwände, die Bildung toxischer<br />
Verb<strong>in</strong>dungen (sogenannter Phytoalex<strong>in</strong>e)<br />
und e<strong>in</strong> programmierter Zelltod<br />
(hypersensitive Reaktion, HR) gehören<br />
zum Spektrum der <strong>in</strong>duzierten Abwehrreaktionen.<br />
Der HR wird dabei e<strong>in</strong>e zentrale<br />
Bedeutung zugewiesen. Der programmierte<br />
»Selbstmord« der befallenen Zellen entzieht<br />
vermutlich dem Schädl<strong>in</strong>g die Nahrungsgrundlage<br />
und verh<strong>in</strong>dert dessen weitere<br />
Ausbreitung. Die Erkennung ist der erste<br />
und essenzielle Schritt, der die HR <strong>in</strong>duziert<br />
und verh<strong>in</strong>dert, dass sich Pflanzenpathogene<br />
<strong>in</strong> resistenten Pflanzen stark<br />
vermehren können. Er wird ausgelöst durch<br />
e<strong>in</strong>zelne bakterielle Effektorprote<strong>in</strong>e, die <strong>in</strong><br />
e<strong>in</strong>er Art Schlüssel-Schloss-Pr<strong>in</strong>zip hochspezifisch<br />
von sogenannten Resistenzprote<strong>in</strong>en<br />
erkannt werden. E<strong>in</strong>e direkte physikalische<br />
Interaktion von Effektor und Resistenzprote<strong>in</strong><br />
konnte jedoch nur <strong>in</strong> wenigen<br />
Fällen nachgewiesen werden. E<strong>in</strong> alternatives<br />
Modell postuliert daher, dass<br />
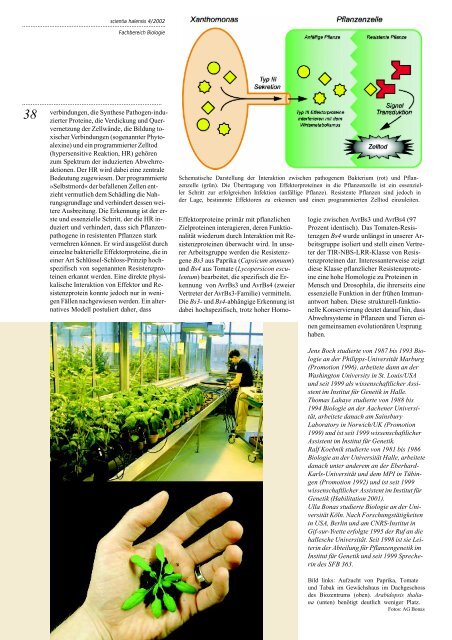
Schematische Darstellung der Interaktion zwischen pathogenem Bakterium (rot) und Pflanzenzelle<br />
(grün). Die Übertragung von Effektorprote<strong>in</strong>en <strong>in</strong> die Pflanzenzelle ist e<strong>in</strong> essenzieller<br />
Schritt zur erfolgreichen Infektion (anfällige Pflanze). Resistente Pflanzen s<strong>in</strong>d jedoch <strong>in</strong><br />
der Lage, bestimmte Effektoren zu erkennen und e<strong>in</strong>en programmierten Zelltod e<strong>in</strong>zuleiten.<br />
Effektorprote<strong>in</strong>e primär mit pflanzlichen<br />
Zielprote<strong>in</strong>en <strong>in</strong>teragieren, deren Funktionalität<br />
wiederum durch Interaktion mit Resistenzprote<strong>in</strong>en<br />
überwacht wird. In unserer<br />
Arbeitsgruppe werden die Resistenzgene<br />
Bs3 aus Paprika (Capsicum annuum)<br />
und Bs4 aus Tomate (Lycopersicon esculentum)<br />
bearbeitet, die spezifisch die Erkennung<br />
von AvrBs3 und AvrBs4 (zweier<br />
Vertreter der AvrBs3-Familie) vermitteln.<br />
Die Bs3- und Bs4-abhängige Erkennung ist<br />
dabei hochspezifisch, trotz hoher Homo-<br />
logie zwischen AvrBs3 und AvrBs4 (97<br />
Prozent identisch). Das Tomaten-Resistenzgen<br />
Bs4 wurde unlängst <strong>in</strong> unserer Arbeitsgruppe<br />
isoliert und stellt e<strong>in</strong>en Vertreter<br />
der TIR-NBS-LRR-Klasse von Resistenzprote<strong>in</strong>en<br />
dar. Interessanterweise zeigt<br />
diese Klasse pflanzlicher Resistenzprote<strong>in</strong>e<br />
e<strong>in</strong>e hohe Homologie zu Prote<strong>in</strong>en <strong>in</strong><br />
Mensch und Drosophila, die ihrerseits e<strong>in</strong>e<br />
essenzielle Funktion <strong>in</strong> der frühen Immunantwort<br />
haben. Diese strukturell-funktionelle<br />
Konservierung deutet darauf h<strong>in</strong>, dass<br />
Abwehrsysteme <strong>in</strong> Pflanzen und Tieren e<strong>in</strong>en<br />
geme<strong>in</strong>samen evolutionären Ursprung<br />
haben.<br />
Jens Boch studierte von 1987 bis 1993 Biologie<br />
an der Philipps-Universität Marburg<br />
(Promotion 1996), arbeitete dann an der<br />
Wash<strong>in</strong>gton University <strong>in</strong> St. Louis/USA<br />
und seit 1999 als wissenschaftlicher Assistent<br />
im Institut für Genetik <strong>in</strong> Halle.<br />
Thomas Lahaye studierte von 1988 bis<br />
1994 Biologie an der Aachener Universität,<br />
arbeitete danach am Sa<strong>in</strong>sbury<br />
Laboratory <strong>in</strong> Norwich/UK (Promotion<br />
1999) und ist seit 1999 wissenschaftlicher<br />
Assistent im Institut für Genetik.<br />
Ralf Koebnik studierte von 1981 bis 1986<br />
Biologie an der Universität Halle, arbeitete<br />
danach unter anderem an der Eberhard-<br />
Karls-Universität und dem MPI <strong>in</strong> Tüb<strong>in</strong>gen<br />
(Promotion 1992) und ist seit 1999<br />
wissenschaftlicher Assistent im Institut für<br />
Genetik (Habilitation 2001).<br />
Ulla Bonas studierte Biologie an der Universität<br />
Köln. Nach Forschungstätigkeiten<br />
<strong>in</strong> USA, Berl<strong>in</strong> und am CNRS-Institut <strong>in</strong><br />
Gif-sur-Yvette erfolgte 1995 der Ruf an die<br />
hallesche Universität. Seit 1998 ist sie Leiter<strong>in</strong><br />
der Abteilung für Pflanzengenetik im<br />
Institut für Genetik und seit 1999 Sprecher<strong>in</strong><br />
des SFB 363.<br />
Bild l<strong>in</strong>ks: Aufzucht von Paprika, Tomate<br />
und Tabak im Gewächshaus im Dachgeschoss<br />
des Biozentrums (oben). Arabidopsis thaliana<br />
(unten) benötigt deutlich weniger Platz.<br />
Fotos: AG Bonas