Einfluss von Mutationen auf die NS2/3-Prozessierung und die ...
Einfluss von Mutationen auf die NS2/3-Prozessierung und die ...
Einfluss von Mutationen auf die NS2/3-Prozessierung und die ...
Sie wollen auch ein ePaper? Erhöhen Sie die Reichweite Ihrer Titel.
YUMPU macht aus Druck-PDFs automatisch weboptimierte ePaper, die Google liebt.
ERGEBNISSE<br />
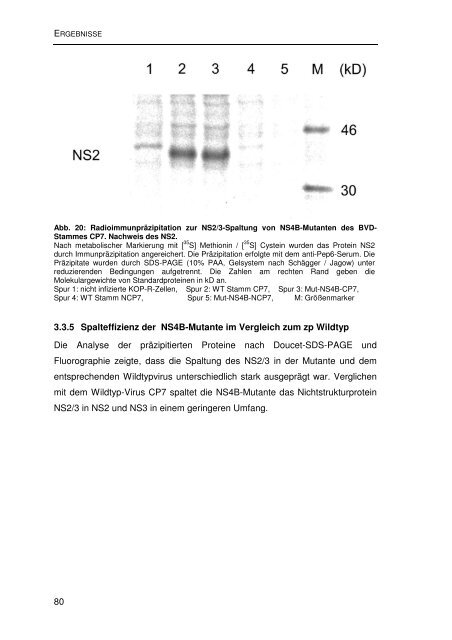
Abb. 20: Radioimmunpräzipitation zur <strong>NS2</strong>/3-Spaltung <strong>von</strong> NS4B-Mutanten des BVD-<br />
Stammes CP7. Nachweis des <strong>NS2</strong>.<br />
Nach metabolischer Markierung mit [ 35 S] Methionin / [ 35 S] Cystein wurden das Protein <strong>NS2</strong><br />
durch Immunpräzipitation angereichert. Die Präzipitation erfolgte mit dem anti-Pep6-Serum. Die<br />
Präzipitate wurden durch SDS-PAGE (10% PAA, Gelsystem nach Schägger / Jagow) unter<br />
reduzierenden Bedingungen <strong>auf</strong>getrennt. Die Zahlen am rechten Rand geben <strong>die</strong><br />
Molekulargewichte <strong>von</strong> Standardproteinen in kD an.<br />
Spur 1: nicht infizierte KOP-R-Zellen, Spur 2: WT Stamm CP7, Spur 3: Mut-NS4B-CP7,<br />
Spur 4: WT Stamm NCP7, Spur 5: Mut-NS4B-NCP7, M: Größenmarker<br />
3.3.5 Spalteffizienz der NS4B-Mutante im Vergleich zum zp Wildtyp<br />
Die Analyse der präzipitierten Proteine nach Doucet-SDS-PAGE <strong>und</strong><br />
Fluorographie zeigte, dass <strong>die</strong> Spaltung des <strong>NS2</strong>/3 in der Mutante <strong>und</strong> dem<br />
entsprechenden Wildtypvirus unterschiedlich stark ausgeprägt war. Verglichen<br />
mit dem Wildtyp-Virus CP7 spaltet <strong>die</strong> NS4B-Mutante das Nichtstrukturprotein<br />
<strong>NS2</strong>/3 in <strong>NS2</strong> <strong>und</strong> NS3 in einem geringeren Umfang.<br />
80