HerzSupplement - Pentalong von Actavis
HerzSupplement - Pentalong von Actavis
HerzSupplement - Pentalong von Actavis
Sie wollen auch ein ePaper? Erhöhen Sie die Reichweite Ihrer Titel.
YUMPU macht aus Druck-PDFs automatisch weboptimierte ePaper, die Google liebt.
a<br />
Relative Luciferaseaktivität<br />
[% <strong>von</strong> Kontrolle]<br />
150<br />
100<br />
50<br />
0<br />
Kontrolle/EtOH<br />
Kontrolle/DMSO<br />
* **<br />
3UTR/EtOH<br />
3UTR/DMSO<br />
Oligonukleotide (ODN),<br />
die zu allen möglichen<br />
Transkripten homolog sind<br />
Annotation<br />
der Gene<br />
(Panther)<br />
SAM-Analyse<br />
(Tiger-MEV)<br />
Aufbringen der<br />
ODN auf<br />
Glasträger<br />
„Heat-Map“<br />
Behandelt<br />
Intensitätsverteilung<br />
Unbehandelt<br />
Herz 35 · 2010 · Supplement II © Urban & Vogel<br />
b<br />
Relative Luciferaseaktivität<br />
[% <strong>von</strong> EtOH bzw. DMSO]<br />
1200<br />
800<br />
100<br />
0<br />
EtOH<br />
RNA <strong>von</strong><br />
unbehandelten<br />
Kontrollen<br />
Alexa<br />
546<br />
ns<br />
0 300 600 800 1200 1600 1800 2400 2600 2800 3000<br />
3200 3600 3800 4000 4200 4800 5000 5400<br />
SV40 late poly(A) signal<br />
NTG<br />
Alexa 546 Alexa 647<br />
Normalisierung<br />
(TIGR-MIDAS)<br />
RNA <strong>von</strong><br />
behandelten<br />
Proben<br />
Fuoreszenz-<br />
Markierung in der<br />
RT-Reaktion<br />
Pseudocoloured<br />
Alexa<br />
647<br />
DMSO<br />
Mischen und<br />
Hybridisierung<br />
*<br />
PETN<br />
Auslesen der<br />
Fluoreszenz-Signale<br />
(Scanarray-Express)<br />
Organische Nitrate<br />
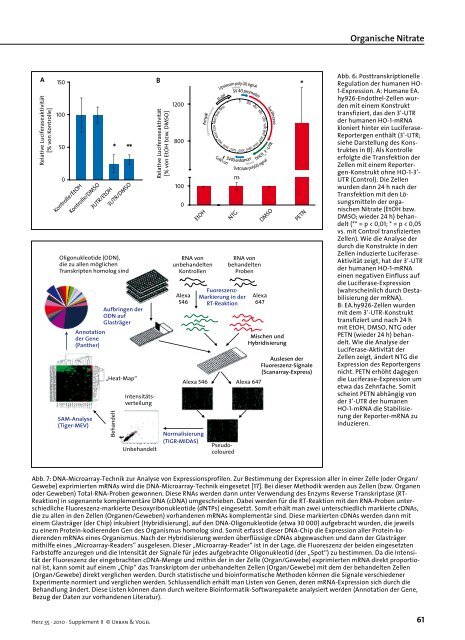
Abb. 6: Posttranskriptionelle<br />
Regulation der humanen HO-<br />
1-Expression. A: Humane EA.<br />
hy926-Endothel-Zellen wurden<br />
mit einem Konstrukt<br />
transfiziert, das den 3’-UTR<br />
der humanen HO-1-mRNA<br />
kloniert hinter ein Luciferase-<br />
Reportergen enthält (3’-UTR;<br />
siehe Darstellung des Konstruktes<br />
in B). Als Kontrolle<br />
erfolgte die Transfektion der<br />
Zellen mit einem Reportergen-Konstrukt<br />
ohne HO-1-3’-<br />
UTR (Control). Die Zellen<br />
wurden dann 24 h nach der<br />
Transfektion mit den Lösungsmitteln<br />
der organischen<br />
Nitrate (EtOH bzw.<br />
DMSO; wieder 24 h) behandelt<br />
(** = p < 0,01; * = p < 0,05<br />
vs. mit Control transfizierten<br />
Zellen). Wie die Analyse der<br />
durch die Konstrukte in den<br />
Zellen induzierte Luciferase-<br />
Aktivität zeigt, hat der 3’-UTR<br />
der humanen HO-1-mRNA<br />
einen negativen Einfluss auf<br />
die Luciferase-Expression<br />
(wahrscheinlich durch Destabilisierung<br />
der mRNA).<br />
B: EA.hy926-Zellen wurden<br />
mit dem 3’-UTR-Konstrukt<br />
transfiziert und nach 24 h<br />
mit EtOH, DMSO, NTG oder<br />
PETN (wieder 24 h) behandelt.<br />
Wie die Analyse der<br />
Luciferase-Aktivität der<br />
Zellen zeigt, ändert NTG die<br />
Expression des Reportergens<br />
nicht. PETN erhöht dagegen<br />
die Luciferase-Expression um<br />
etwa das Zehnfache. Somit<br />
scheint PETN abhängig <strong>von</strong><br />
der 3’-UTR der humanen<br />
HO-1-mRNA die Stabilisierung<br />
der Reporter-mRNA zu<br />
induzieren.<br />
Abb. 7: DNA-Microarray-Technik zur Analyse <strong>von</strong> Expressionsprofilen. Zur Bestimmung der Expression aller in einer Zelle (oder Organ/<br />
Gewebe) exprimierten mRNAs wird die DNA-Microarray-Technik eingesetzt [17]. Bei dieser Methodik werden aus Zellen (bzw. Organen<br />
oder Geweben) Total-RNA-Proben gewonnen. Diese RNAs werden dann unter Verwendung des Enzyms Reverse Transkriptase (RT-<br />
Reaktion) in sogenannte komplementäre DNA (cDNA) umgeschrieben. Dabei werden für die RT-Reaktion mit den RNA-Proben unterschiedliche<br />
Fluoreszenz-markierte Desoxyribonukleotide (dNTPs) eingesetzt. Somit erhält man zwei unterschiedlich markierte cDNAs,<br />
die zu allen in den Zellen (Organen/Geweben) vorhandenen mRNAs komplementär sind. Diese markierten cDNAs werden dann mit<br />
einem Glasträger (der Chip) inkubiert (Hybridisierung), auf den DNA-Oligonukleotide (etwa 30 000) aufgebracht wurden, die jeweils<br />
zu einem Protein-kodierenden Gen des Organismus homolog sind. Somit erfasst dieser DNA-Chip die Expression aller Protein-kodierenden<br />
mRNAs eines Organismus. Nach der Hybridisierung werden überflüssige cDNAs abgewaschen und dann der Glasträger<br />
mithilfe eines „Microarray-Readers“ ausgelesen. Dieser „Microarray-Reader“ ist in der Lage, die Fluoreszenz der beiden eingesetzten<br />
Farbstoffe anzuregen und die Intensität der Signale für jedes aufgebrachte Oligonukleotid (der „Spot“) zu bestimmen. Da die Intensität<br />
der Fluoreszenz der eingebrachten cDNA-Menge und mithin der in der Zelle (Organ/Gewebe) exprimierten mRNA direkt proportional<br />
ist, kann somit auf einem „Chip“ das Transkriptom der unbehandelten Zellen (Organ/Gewebe) mit dem der behandelten Zellen<br />
(Organ/Gewebe) direkt verglichen werden. Durch statistische und bioinformatische Methoden können die Signale verschiedener<br />
Experimente normiert und verglichen werden. Schlussendlich erhält man Listen <strong>von</strong> Genen, deren mRNA-Expression sich durch die<br />
Behandlung ändert. Diese Listen können dann durch weitere Bioinformatik-Softwarepakete analysiert werden (Annotation der Gene,<br />
Bezug der Daten zur vorhandenen Literatur).<br />
61