- Page 2 and 3:
BENJAMIN LEWIN Undcrgrad u :m~ and
- Page 4 and 5:
Prefácio A ciência é uma maravi1
- Page 6 and 7:
Sumário 1 Os Genes São DNA 1 18 R
- Page 8 and 9:
38 CAPinJLO 3 O GfnE IIITERROHPIOO
- Page 10 and 11:
40 CAPITULO 3 o G!N[ INT!RROMPIDO ~
- Page 12 and 13:
42 CAPÍTULO 3 O GENE INTERROMPIDO
- Page 14 and 15:
44 CAPITuLO 3 O ~ENE INTERROMPIOO 6
- Page 16 and 17:
46 CAPITULO 3 O GEkE OOERROMP!OO Pr
- Page 18 and 19:
48 CAPtTULO 3 O GIME IHTlRROMPIDO E
- Page 20 and 21:
50 CAPÍTULO 3 O GENE INIERRO~PIDO
- Page 22 and 23:
52 CAPÍTUlO 3 O GEM INTERROMPIDO U
- Page 24 and 25:
54 CAPÍTULO 3 O GENE INTERRO~PIOO
- Page 26 and 27:
56 CAPITuLO 4 O CIIHTEOOO 00 6IHOII
- Page 28 and 29:
58 CAPÍTULO 4 O CONTEÚOO 00 GENOM
- Page 30 and 31:
60 CAPÍTULO 4 O CONTEUOO 00 GEN()j
- Page 32 and 33:
62 CAPÍTULO ' O tONTEUOO 00 ~ENOMA
- Page 34 and 35:
64 CAPÍTULO 4 O CONTEUOO 00 GEHOMA
- Page 36 and 37:
66 CAPÍTULO 4 O COHTEÚOO 00 GENOM
- Page 38 and 39:
68 CAPÍTULO ' O COIIIEÚOO 00 6EkO
- Page 40 and 41:
70 CAPITuLO 4 O (OiffiliDO 00 G!M)H
- Page 42 and 43:
72 CAPÍTULO 4 O CONITÚOO DO GENOM
- Page 44 and 45:
74 CAPITULO 4 O CONTEÚDO DO GEHDMA
- Page 46 and 47:
.U Introdução .a A Duplicação G
- Page 48 and 49:
100 CAPÍTULO 6 M;RIJPAIWS relacion
- Page 50 and 51:
102 CAPiTUlO 6 AGRUPAIIEHTOS E REPr
- Page 52 and 53:
104 CAPITuLO 6 AGRUPAMENTOS E REPrn
- Page 54 and 55:
106 CAPÍTULO 6 AGR UPAMEIITOS E RE
- Page 56 and 57:
108 CAPÍTULO 6 AGRUPAMENTOS E REPE
- Page 58 and 59:
110 CAPITulO 6 AGiUPAKEHIOS! R!Kn(
- Page 60 and 61:
112 CAPÍTULO 6 AGRUPAM!IaOS E REPI
- Page 62 and 63:
f 6.1 A transcrfçJo de agrupamento
- Page 64 and 65:
116 CAPÍTULO 6 AGRUPAMENlOS E REPE
- Page 66 and 67:
118 CAPITULO 6 AGQUPAMt'ITOS E liPr
- Page 68 and 69:
120 CAPiTUlO 6 AGRUPAIIENTOS E iJPf
- Page 70 and 71:
122 CAPITuLO 6 AGRUPAMENTOS E REPEn
- Page 72 and 73:
124 CAPiTULO 6 AGRUPAMERTOS E REPEn
- Page 74 and 75:
126 CAPfTULO 6 AGRUPAJ(EHTOS E REPE
- Page 76 and 77:
5.2 O NUMERO OE GENES E l~ BACTÉRI
- Page 78 and 79:
5.3 O NÚ I~ERO OE GENES É CONHECI
- Page 80 and 81:
5. ~ QUANTOS TIPOS DIFERENTES OE GE
- Page 82 and 83:
5.5 O GENOMA HU~IANO TEM ~I E N OS
- Page 84 and 85:
5.6 COMO OS GENES E OUTRAS SEQÜ1NO
- Page 86 and 87:
5.8 ESPÉCIES MAIS COMPLEXAS EVOLUE
- Page 88 and 89:
5.9 QUANTOS GENES SÃO ESSENCIAIS?
- Page 90 and 91:
5.9 QUANTOS GENES SÃO ESSENCIAIS?
- Page 92 and 93:
5.12 O NÚMERO OE GENES EXPRESSOS P
- Page 94 and 95:
REFERÊNCIAS 95 Resumo Os gcnomas s
- Page 96 and 97:
REFERENCIAS 97 O Cromossomo Y Possu
- Page 98 and 99:
128 CAPITULO 7 ORNA MENSAGEIRO IJID
- Page 100 and 101:
130 CAPÍTULO 7 ORNA MENSAGEIRO O t
- Page 102 and 103:
13 2 CAPITULO 7 O IlHA MlliSAGORO T
- Page 104 and 105:
134 CAPÍTULO 7 O lli ••••
- Page 106 and 107:
136 CAPÍTULO 7 O RNA MENSAGEIRO fl
- Page 108 and 109:
138 CAPÍTULO 7 O RIIA MEIISAGE!RO
- Page 110 and 111:
140 CAPITULO 7 . ORNA MENSAGURO A m
- Page 112 and 113:
142 CAPÍTUlO 7, O RHA MENSAGEIRO ~
- Page 114 and 115:
144 CAPITulO 1 O RHA IIEl
- Page 116 and 117:
146 CAPITULO 7 O RHA IIENSAGE!Jt()
- Page 118 and 119:
148 CAPiTULO 7 ORNA ~ENSAGEJ RO dem
- Page 120 and 121:
150 CAPtfuLO 7 O ~'IA K!NSAGURO B M
- Page 122 and 123:
522 CAPÍTULO 21 TRANsroiONS • A
- Page 124 and 125:
J - - · •• -------·- -· •
- Page 126 and 127:
526 CAPITULO 21 TR4NSP050NS Element
- Page 128 and 129:
.., 528 CAPITULO 21 TRANSPOSONS O t
- Page 130 and 131:
530 CAPfTULO 21 TRANSPOSONS IJH.M I
- Page 132 and 133:
532 CAPÍTULO 21 TRA~SPOSONS Transp
- Page 134 and 135:
534 CAPÍTULO 21 IRANSPOSONS í Tn5
- Page 136 and 137:
536 CAPÍTULO 21 Tl!ANSPOSONS corHe
- Page 138 and 139:
538 CAPITULO 21 TRA.~SPOSO.~S rem r
- Page 140 and 141:
540 CAPITulO 21 IRAIISPOSOiiS ._ç
- Page 142 and 143:
542 CAPITULO 21 TRAHII'OSOHS se ama
- Page 144 and 145:
se conhece a natureza dos evenros r
- Page 146 and 147:
cisa (um evenro incomum em ouuos si
- Page 148 and 149:
548 CAPÍTULO 21 TRAHSPOSONS Refer
- Page 150 and 151:
fnD Introdução fiJfJ O Ciclo de V
- Page 152 and 153:
552 CAPÍTULO 22 RETR()VÍRUS E RET
- Page 154 and 155:
554 CAPÍTULO 22 RETROViRUS E RETRO
- Page 156 and 157:
556 CAPtruLO 22 RETRO'IÍAUS E AETR
- Page 158 and 159:
55! CA?:W..O 22 ,.,,, R
- Page 160 and 161:
~ W!nJLO ZZ RETROviRUS E RETROIRANS
- Page 162 and 163:
562 CAPÍTULO 22 RETl
- Page 164 and 165:
564 CAPiTULO 22 REJROYÍRUS E RETRQ
- Page 166 and 167:
própria rransposiçlo. nw também
- Page 168 and 169:
568 CAPÍTULO 22 RElROYIRUS E RETRO
- Page 170 and 171:
IBI Introdução 1m Os Replicons Po
- Page 172 and 173:
378 CAPÍTULO 15 O REPLICON gadas,
- Page 174 and 175:
380 CAPÍTULO 15 O REPLICON Y simpl
- Page 176 and 177:
382 CAPÍTULO 15 O REPLICON O inibi
- Page 178 and 179:
384 CAPÍTULO 15 O REPLICON FIGURA
- Page 180 and 181:
386 CAPÍTULO 15 O REPUCON . .. . ~
- Page 182 and 183:
388 CAPÍTULO 15 O REPLICON minados
- Page 184 and 185:
390 CAPÍTULO 15 O REPLICON plicaç
- Page 186 and 187:
IDJ Introdução 1nB As DNA Polimer
- Page 188 and 189:
430 CAPÍTULO 18 REPLICAÇÃO DE DN
- Page 190 and 191:
43 2 CAPÍTULO 18 REPLICAÇÃO DE D
- Page 192 and 193:
434 CAPÍTULO 18 REPLICAÇÃO DE DN
- Page 194 and 195:
m~CAÇÃO DE DNA A helicase circund
- Page 196 and 197:
438 CAPÍTULO 18 REPliCAÇÃO DE DN
- Page 198 and 199:
440 CAPÍTU LO 18 REPLICAÇÃO DE D
- Page 200 and 201:
442 CAPÍTULO 18 REPLICAÇÃO OE ON
- Page 202 and 203:
444 CAPÍTULO 18 REPLICAÇÃO DE DN
- Page 204 and 205:
1 446 CAPÍTULO 18 REPLICAÇÃO DE
- Page 206 and 207:
448 CAPÍTULO 18 REPLICAÇÃO DE ON
- Page 208 and 209:
M •• - ... ~- .... ~ - - -; ~ 4
- Page 210 and 211:
452 CAPÍTULO 18 REPLICAÇÃO DE DN
- Page 212 and 213:
454 CAPÍTULO 18 REPUCAÇÃO DE DNA
- Page 214 and 215:
í 456 CAPÍTULO 18 REPLICAÇÃO DE
- Page 216 and 217:
CAPÍTULO 11 TRANSCRIÇÃO 257 •
- Page 218 and 219:
11.2 A TRANSCRIÇÃO OCORRE COM O P
- Page 220 and 221:
11.4 A RNA POLIMERASE DO FAGO T7 É
- Page 222 and 223:
11.5 A ESTRUTURA CRISTALINA SUGERE
- Page 224 and 225:
11.6 A RNA POLIMERASE BACTERIANA CO
- Page 226 and 227:
11.8 A ASSOCIAÇÃO COM O FATOR SIG
- Page 228 and 229:
11.9 UMA RNA POLIMERASE PARADA PODE
- Page 230 and 231:
11.11 O FATOR SIGMA CONTROLA A LIGA
- Page 232 and 233:
11.12 O RECONHECIMENTO DO PROMOTOR
- Page 234 and 235:
11.14 A RNA POLIMERASE LIGA-SE A UM
- Page 236 and 237:
,- 11.15 O SUPERENOVELAMENTO É UMA
- Page 238 and 239:
11.16 A SUBSTITUIÇÃO DE FATORES S
- Page 240 and 241:
11.17 O FATOR SIGMA ESTABELECE CONT
- Page 242 and 243:
11.19 A ESPORULAÇÃO É CONTROLADA
- Page 244 and 245:
11.19 A ESPORULAÇÃO É CONTROLADA
- Page 246 and 247:
11.21 EXISTEM DOIS TIPOS DE TERMINA
- Page 248 and 249:
11.22 COMO O FATOR Rho ATUA? 289 li
- Page 250 and 251:
11.23 A ANTITERMINAÇÃO É UM EVEN
- Page 252 and 253:
11.25 OS FATORES DE TERMINAÇÃO E
- Page 254 and 255:
11. 26 RESUMO 295 l sas em todos os
- Page 256 and 257:
REFERÊNCIAS 297 Cramer, P., Bushne
- Page 258 and 259:
REFERÊNCIAS 299 Gcisclmann,]., \Xf
- Page 260 and 261:
152 CAPÍTULO 8 SÍNTESE PROTÉICA
- Page 262 and 263:
154 CA?~LO 8 SÍNTESE PROTÉICA Có
- Page 264 and 265:
156 CAPÍTULO 8 SÍNTESE PROTÉICA
- Page 266 and 267:
158 CAPÍTULO 8 SÍNTESE PROTÉICA
- Page 268 and 269:
- O 160 CAPÍTULO 8 SÍNTESE PROTÉ
- Page 270 and 271:
162 CAPÍTUlO 8 SÍNTESE PROTÉICA
- Page 272 and 273:
164 CAPÍTULO 8 SÍNTESE PROTÉICA
- Page 274 and 275:
166 CAPÍTULO 8 SÍNTESE PROTÉICA
- Page 276 and 277:
168 CAPÍTULO 8 SÍNTESE PROTÉICA
- Page 278 and 279:
170 CAPÍTULO 8 SÍNTESE PROTÉICA
- Page 280 and 281:
172 CAPÍTULO 8 SÍNTESE PROTÉICA
- Page 282 and 283: 174 CAPÍTULO 8 SÍNTESE PROTÉICA
- Page 284 and 285: 17 6 CAPÍTULO 8 SÍNTESE PROTÉICA
- Page 286 and 287: 178 CAPÍTULO 8 SÍNTESE PROTÉICA
- Page 288 and 289: 180 CAPÍTULO 8 SÍNTESE PROTÉICA
- Page 290 and 291: 182 CAPÍTULO 8 SÍNTESE PROTÉICA
- Page 292 and 293: 184 CAPÍTULO 8 SÍNTESE PROTÉICA
- Page 294 and 295: 186 CAPÍTULO 8 SÍNTESE PROTÉICA
- Page 296 and 297: 188 CAPÍTULO 8 SÍNTESE PROTÉICA
- Page 298 and 299: 33 2 CAPÍTULO 13 O RNA REGULA TÓR
- Page 300 and 301: 334 CAPÍTULO 13 O RNA REG ULATÓRI
- Page 302 and 303: 336 CAPÍTULO 13 O RNA REG ULATÓRI
- Page 304 and 305: 338 CAPÍTULO 13 O RNA REGULATÓRIO
- Page 306 and 307: 340 CAPÍTULO 13 ORNA REG ULATÓRIO
- Page 308 and 309: 342 CAPÍTULO 13 O RNA REGULATÓRIO
- Page 310 and 311: A .. 344 CAPÍTULO 13 ORNA REGULAT
- Page 312 and 313: 346 CAPÍTULO 13 O RNA REGULATÓRIO
- Page 314 and 315: 348 CAPÍTULO 13 O RNA REGULATÓRIO
- Page 316 and 317: 610 CAPÍTULO 24 PROMOTORES E INTEN
- Page 318 and 319: 612 CAPÍTULO 24 PROMOTORES E INTEN
- Page 320 and 321: 614 CAPÍTULO 24 PROHOTORES E INTEN
- Page 322 and 323: 616 CAPÍTULO 24 PROMOTORES E JNTEN
- Page 324 and 325: 618 CAPÍTULO 24 PROMOTORES E INTEN
- Page 326 and 327: 620 CAPÍTU LO 24 PROMOTORES E INTE
- Page 328 and 329: 622 CAPÍTULO 24 PROMOTORES E INTEN
- Page 330 and 331: 624 CAPÍTULO 24 PROMOTORES E INTEN
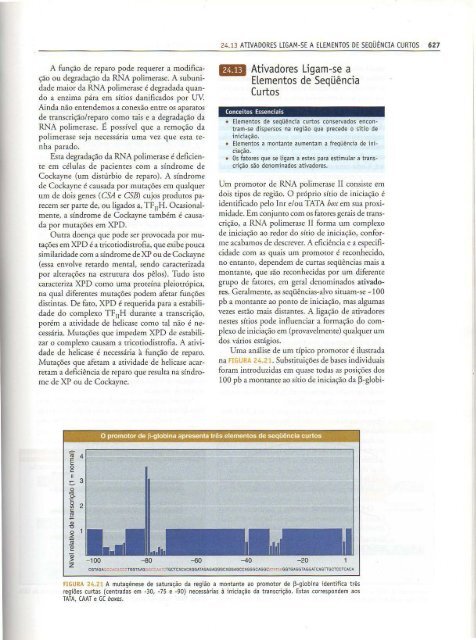
- Page 334 and 335: 628 CAPÍTULO 24 PROt·IOTORES E IN
- Page 336 and 337: 630 CAPÍTULO 24 PROMOTORES E INTEN
- Page 338 and 339: 632 CAPÍTULO 24 PROMOTORES E INTEN
- Page 340 and 341: é3 WITULO 24 PROMOTORES E INTENSIF
- Page 342 and 343: 636 ::;.::lf""u-0 24 í'ilOMOTORES
- Page 344 and 345: 638 CAPÍTULO 24 PROMOTORES E INTEN
- Page 346 and 347: ImO Introdução lmll Junções de
- Page 348 and 349: 26.1 I NTRODUÇÃO 669 fliJfB A Cli
- Page 350 and 351: 26.3 AS JUNÇÕ ES DE SPUCING SÃO
- Page 352 and 353: 26.4 O SPUCING DO PRÉ-mRNA OCORRE
- Page 354 and 355: 26.5 snRNAs SÃO NECESSÁRIOS PARA
- Page 356 and 357: 26.6 A sn RNP Ul INICIA O SPUCING 6
- Page 358 and 359: 26.8 CINCO snRNPS FORMAM O SPUCEOSS
- Page 360 and 361: 26.9 UM APARATO ALTERNATIVO DE SPUC
- Page 362 and 363: 26.11 ÍNTRONS DO GRUPO II SOFREM A
- Page 364 and 365: 26.12 O SPLICING ALTERNATIVO ENVOLV
- Page 366 and 367: esulrado do spficing alternativo, p
- Page 368 and 369: 26 .13 REAÇÕES DE TRANS-SPLICING
- Page 370 and 371: Incubação com o extrato de spli=t
- Page 372 and 373: •au A Resposta à Proteína Nãoe
- Page 374 and 375: 26.19 AS EXTREMIDADES 3' DOS mRNAs
- Page 376 and 377: 26.21 A SÍNTESE DE rRNA REQUER EVE
- Page 378 and 379: tMij Pequenos RNAs São Necessário
- Page 380 and 381: REFERÊNCIAS 701 splicing. As snRNP
- Page 382 and 383:
REFERÊNCIAS 703 Luo, M. L., Zhou,
- Page 384 and 385:
REFERÊNCIAS 705 Ganor, P., Caizerg
- Page 386 and 387:
846 GLOSSÁRIO O anticódon é uma
- Page 388 and 389:
848 GLOSSÁRIO Círculo rolante é
- Page 390 and 391:
850 GLOSSÁRIO Desoxirribonudease
- Page 392 and 393:
852 GLOSSÁRIO com um códon de ini
- Page 394 and 395:
854 GLOSSÁRIO A hibridização irt
- Page 396 and 397:
856 GLOSSÁRIO Um lipopolissacaríd
- Page 398 and 399:
858 GLOSSÁRIO Opina é um derivado
- Page 400 and 401:
860 GLOSSÁRIO Uma proteína transm
- Page 402 and 403:
862 GLOSSÁRIO rradução) pela lig
- Page 404 and 405:
864 GLOSSÁRIO O sício de ramifica
- Page 406 and 407:
866 GLOSSÁRIO um canal, permitindo