Untersuchungen zum Argininstoffwechsel bei Mycobacterium bovis ...
Untersuchungen zum Argininstoffwechsel bei Mycobacterium bovis ...
Untersuchungen zum Argininstoffwechsel bei Mycobacterium bovis ...
Sie wollen auch ein ePaper? Erhöhen Sie die Reichweite Ihrer Titel.
YUMPU macht aus Druck-PDFs automatisch weboptimierte ePaper, die Google liebt.
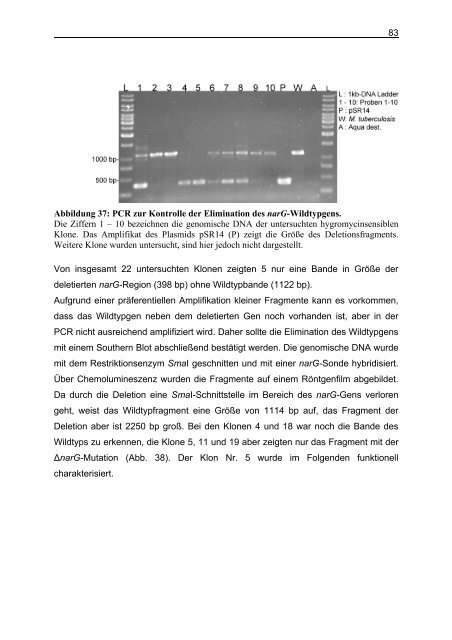
Abbildung 37: PCR zur Kontrolle der Elimination des narG-Wildtypgens.<br />
Die Ziffern 1 – 10 bezeichnen die genomische DNA der untersuchten hygromycinsensiblen<br />
Klone. Das Amplifikat des Plasmids pSR14 (P) zeigt die Größe des Deletionsfragments.<br />
Weitere Klone wurden untersucht, sind hier jedoch nicht dargestellt.<br />
Von insgesamt 22 untersuchten Klonen zeigten 5 nur eine Bande in Größe der<br />
deletierten narG-Region (398 bp) ohne Wildtypbande (1122 bp).<br />
Aufgrund einer präferentiellen Amplifikation kleiner Fragmente kann es vorkommen,<br />
dass das Wildtypgen neben dem deletierten Gen noch vorhanden ist, aber in der<br />
PCR nicht ausreichend amplifiziert wird. Daher sollte die Elimination des Wildtypgens<br />
mit einem Southern Blot abschließend bestätigt werden. Die genomische DNA wurde<br />
mit dem Restriktionsenzym SmaI geschnitten und mit einer narG-Sonde hybridisiert.<br />
Über Chemolumineszenz wurden die Fragmente auf einem Röntgenfilm abgebildet.<br />
Da durch die Deletion eine SmaI-Schnittstelle im Bereich des narG-Gens verloren<br />
geht, weist das Wildtypfragment eine Größe von 1114 bp auf, das Fragment der<br />
Deletion aber ist 2250 bp groß. Bei den Klonen 4 und 18 war noch die Bande des<br />
Wildtyps zu erkennen, die Klone 5, 11 und 19 aber zeigten nur das Fragment mit der<br />
∆narG-Mutation (Abb. 38). Der Klon Nr. 5 wurde im Folgenden funktionell<br />
charakterisiert.<br />
83









![Tmnsudation.] - TiHo Bibliothek elib](https://img.yumpu.com/23369022/1/174x260/tmnsudation-tiho-bibliothek-elib.jpg?quality=85)






