Mechanische Anisotropie von Proteinen in ...
Mechanische Anisotropie von Proteinen in ...
Mechanische Anisotropie von Proteinen in ...
Erfolgreiche ePaper selbst erstellen
Machen Sie aus Ihren PDF Publikationen ein blätterbares Flipbook mit unserer einzigartigen Google optimierten e-Paper Software.
52 6. M<strong>in</strong>imalmodell der Mechanik e<strong>in</strong>er Prote<strong>in</strong>struktur<br />
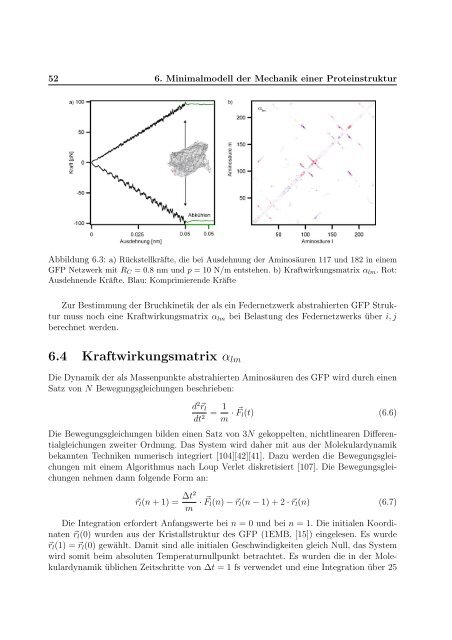
Abbildung 6.3: a) Rückstellkräfte, die bei Ausdehnung der Am<strong>in</strong>osäuren 117 und 182 <strong>in</strong> e<strong>in</strong>em<br />
GFP Netzwerk mit R C = 0.8 nm und p = 10 N/m entstehen. b) Kraftwirkungsmatrix α lm . Rot:<br />
Ausdehnende Kräfte. Blau: Komprimierende Kräfte<br />
Zur Bestimmung der Bruchk<strong>in</strong>etik der als e<strong>in</strong> Federnetzwerk abstrahierten GFP Struktur<br />
muss noch e<strong>in</strong>e Kraftwirkungsmatrix α lm bei Belastung des Federnetzwerks über i, j<br />
berechnet werden.<br />
6.4 Kraftwirkungsmatrix α lm<br />
Die Dynamik der als Massenpunkte abstrahierten Am<strong>in</strong>osäuren des GFP wird durch e<strong>in</strong>en<br />
Satz <strong>von</strong> N Bewegungsgleichungen beschrieben:<br />
d 2 ⃗r l<br />
dt 2 = 1 m · ⃗F l (t) (6.6)<br />
Die Bewegungsgleichungen bilden e<strong>in</strong>en Satz <strong>von</strong> 3N gekoppelten, nichtl<strong>in</strong>earen Differentialgleichungen<br />
zweiter Ordnung. Das System wird daher mit aus der Molekulardynamik<br />
bekannten Techniken numerisch <strong>in</strong>tegriert [104][42][41]. Dazu werden die Bewegungsgleichungen<br />
mit e<strong>in</strong>em Algorithmus nach Loup Verlet diskretisiert [107]. Die Bewegungsgleichungen<br />
nehmen dann folgende Form an:<br />
⃗r l (n + 1) = ∆t2<br />
m · ⃗F l (n) − ⃗r l (n − 1) + 2 · ⃗r l (n) (6.7)<br />
Die Integration erfordert Anfangswerte bei n = 0 und bei n = 1. Die <strong>in</strong>itialen Koord<strong>in</strong>aten<br />
⃗r l (0) wurden aus der Kristallstruktur des GFP (1EMB, [15]) e<strong>in</strong>gelesen. Es wurde<br />
⃗r l (1) = ⃗r l (0) gewählt. Damit s<strong>in</strong>d alle <strong>in</strong>itialen Geschw<strong>in</strong>digkeiten gleich Null, das System<br />
wird somit beim absoluten Temperaturnullpunkt betrachtet. Es wurden die <strong>in</strong> der Molekulardynamik<br />
üblichen Zeitschritte <strong>von</strong> ∆t = 1 fs verwendet und e<strong>in</strong>e Integration über 25