Ataxie mit okulomotorischer Apraxie Typ 2: Charakterisierung des ...
Ataxie mit okulomotorischer Apraxie Typ 2: Charakterisierung des ...
Ataxie mit okulomotorischer Apraxie Typ 2: Charakterisierung des ...
Erfolgreiche ePaper selbst erstellen
Machen Sie aus Ihren PDF Publikationen ein blätterbares Flipbook mit unserer einzigartigen Google optimierten e-Paper Software.
Ergebnisse 36<br />
Tyrosin zu Cystein in der Aminosäure-Sequenz zur Folge (c.971A>G, p.Y324C). Eine<br />
weitere Sequenzveränderung im SETX-Gen wurde bei der Sequenzierung nicht entdeckt.<br />
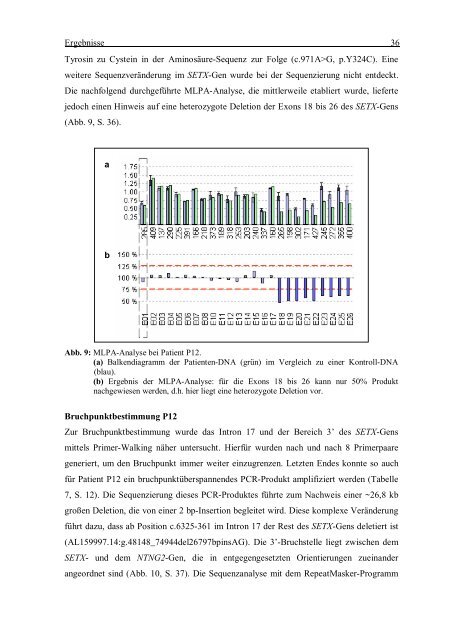
Die nachfolgend durchgeführte MLPA-Analyse, die <strong>mit</strong>tlerweile etabliert wurde, lieferte<br />
jedoch einen Hinweis auf eine heterozygote Deletion der Exons 18 bis 26 <strong>des</strong> SETX-Gens<br />
(Abb. 9, S. 36).<br />
a<br />
b<br />
Abb. 9: MLPA-Analyse bei Patient P12.<br />
(a) Balkendiagramm der Patienten-DNA (grün) im Vergleich zu einer Kontroll-DNA<br />
(blau).<br />
(b) Ergebnis der MLPA-Analyse: für die Exons 18 bis 26 kann nur 50% Produkt<br />
nachgewiesen werden, d.h. hier liegt eine heterozygote Deletion vor.<br />
Bruchpunktbestimmung P12<br />
Zur Bruchpunktbestimmung wurde das Intron 17 und der Bereich 3’ <strong>des</strong> SETX-Gens<br />
<strong>mit</strong>tels Primer-Walking näher untersucht. Hierfür wurden nach und nach 8 Primerpaare<br />
generiert, um den Bruchpunkt immer weiter einzugrenzen. Letzten En<strong>des</strong> konnte so auch<br />
für Patient P12 ein bruchpunktüberspannen<strong>des</strong> PCR-Produkt amplifiziert werden (Tabelle<br />
7, S. 12). Die Sequenzierung dieses PCR-Produktes führte zum Nachweis einer ~26,8 kb<br />
großen Deletion, die von einer 2 bp-Insertion begleitet wird. Diese komplexe Veränderung<br />
führt dazu, dass ab Position c.6325-361 im Intron 17 der Rest <strong>des</strong> SETX-Gens deletiert ist<br />
(AL159997.14:g.48148_74944del26797bpinsAG). Die 3’-Bruchstelle liegt zwischen dem<br />
SETX- und dem NTNG2-Gen, die in entgegengesetzten Orientierungen zueinander<br />
angeordnet sind (Abb. 10, S. 37). Die Sequenzanalyse <strong>mit</strong> dem RepeatMasker-Programm