Untersuchung von Cyclodextrinkomplexen - OPUS - Universität ...
Untersuchung von Cyclodextrinkomplexen - OPUS - Universität ...
Untersuchung von Cyclodextrinkomplexen - OPUS - Universität ...
Sie wollen auch ein ePaper? Erhöhen Sie die Reichweite Ihrer Titel.
YUMPU macht aus Druck-PDFs automatisch weboptimierte ePaper, die Google liebt.
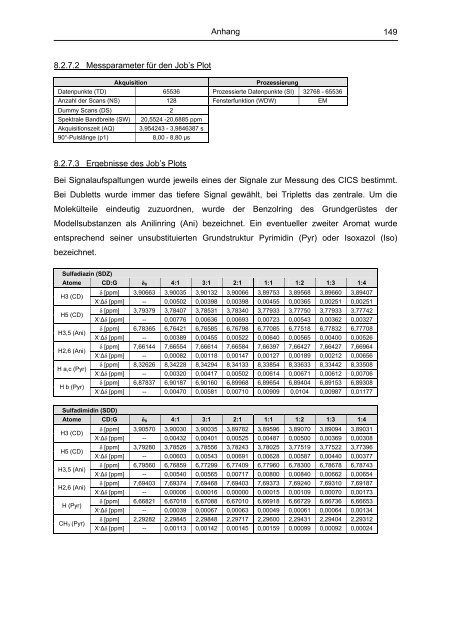
8.2.7.2 Messparameter für den Job’s Plot<br />
Anhang<br />
Akquisition Prozessierung<br />
Datenpunkte (TD) 65536 Prozessierte Datenpunkte (SI) 32768 - 65536<br />
Anzahl der Scans (NS) 128 Fensterfunktion (WDW) EM<br />
Dummy Scans (DS) 2<br />
Spektrale Bandbreite (SW) 20,5524 -20,6885 ppm<br />
Akquisitionszeit (AQ) 3,954243 - 3,9846387 s<br />
90°-Pulslänge (p1) 8,00 - 8,80 µs<br />
8.2.7.3 Ergebnisse des Job’s Plots<br />
Bei Signalaufspaltungen wurde jeweils eines der Signale zur Messung des CICS bestimmt.<br />
Bei Dubletts wurde immer das tiefere Signal gewählt, bei Tripletts das zentrale. Um die<br />
Molekülteile eindeutig zuzuordnen, wurde der Benzolring des Grundgerüstes der<br />
Modellsubstanzen als Anilinring (Ani) bezeichnet. Ein eventueller zweiter Aromat wurde<br />
entsprechend seiner unsubstituierten Grundstruktur Pyrimidin (Pyr) oder Isoxazol (Iso)<br />
bezeichnet.<br />
Sulfadiazin (SDZ)<br />
Atome CD:G δ0 4:1 3:1 2:1 1:1 1:2 1:3 1:4<br />
H3 (CD)<br />
δ [ppm]<br />
X·Δδ [ppm]<br />
3,90663<br />
--<br />
3,90035<br />
0,00502<br />
3,90132<br />
0,00398<br />
3,90066<br />
0,00398<br />
3,89753<br />
0,00455<br />
3,89568<br />
0,00365<br />
3,89660<br />
0,00251<br />
3,89407<br />
0,00251<br />
H5 (CD)<br />
δ [ppm] 3,79379 3,78407 3,78531 3,78340 3,77933 3,77750 3,77933 3,77742<br />
X·Δδ [ppm] -- 0,00776 0,00636 0,00693 0,00723 0,00543 0,00362 0,00327<br />
δ [ppm] 6,78365 6,76421 6,76585 6,76798 6,77085 6,77518 6,77832 6,77708<br />
H3,5 (Ani) X·Δδ [ppm] -- 0,00389 0,00455 0,00522 0,00640 0,00565 0,00400 0,00526<br />
δ [ppm] 7,66144 7,66554 7,66614 7,66584 7,66397 7,66427 7,66427 7,66964<br />
H2,6 (Ani) X·Δδ [ppm] -- 0,00082 0,00118 0,00147 0,00127 0,00189 0,00212 0,00656<br />
δ [ppm] 8,32626 8,34228 8,34294 8,34133 8,33854 8,33633 8,33442 8,33508<br />
H a,c (Pyr) X·Δδ [ppm] -- 0,00320 0,00417 0,00502 0,00614 0,00671 0,00612 0,00706<br />
H b (Pyr)<br />
Sulfadimidin (SDD)<br />
δ [ppm] 6,87837 6,90187 6,90160 6,89968 6,89654 6,89404 6,89153 6,89308<br />
X·Δδ [ppm] -- 0,00470 0,00581 0,00710 0,00909 0,0104 0,00987 0,01177<br />
Atome CD:G δ0 4:1 3:1 2:1 1:1 1:2 1:3 1:4<br />
H3 (CD)<br />
H5 (CD)<br />
δ [ppm] 3,90570 3,90030 3,90035 3,89782 3,89596 3,89070 3,89094 3,89031<br />
X·Δδ [ppm] -- 0,00432 0,00401 0,00525 0,00487 0,00500 0,00369 0,00308<br />
δ [ppm] 3,79280 3,78526 3,78556 3,78243 3,78025 3,77519 3,77522 3,77396<br />
X·Δδ [ppm] -- 0,00603 0,00543 0,00691 0,00628 0,00587 0,00440 0,00377<br />
δ [ppm] 6,79560 6,76859 6,77299 6,77409 6,77960 6,78300 6,78678 6,78743<br />
H3,5 (Ani) X·Δδ [ppm] -- 0,00540 0,00565 0,00717 0,00800 0,00840 0,00662 0,00654<br />
δ [ppm] 7,69403 7,69374 7,69468 7,69403 7,69373 7,69240 7,69310 7,69187<br />
H2,6 (Ani) X·Δδ [ppm] -- 0,00006 0,00016 0,00000 0,00015 0,00109 0,00070 0,00173<br />
H (Pyr)<br />
CH3 (Pyr)<br />
δ [ppm] 6,66821 6,67018 6,67088 6,67010 6,66918 6,66729 6,66736 6,66653<br />
X·Δδ [ppm] -- 0,00039 0,00067 0,00063 0,00049 0,00061 0,00064 0,00134<br />
δ [ppm] 2,29282 2,29845 2,29848 2,29717 2,29600 2,29431 2,29404 2,29312<br />
X·Δδ [ppm] -- 0,00113 0,00142 0,00145 0,00159 0,00099 0,00092 0,00024<br />
149