Pyruvat-Produktion durch acetatauxotrophe - JUWEL ...
Pyruvat-Produktion durch acetatauxotrophe - JUWEL ...
Pyruvat-Produktion durch acetatauxotrophe - JUWEL ...
Erfolgreiche ePaper selbst erstellen
Machen Sie aus Ihren PDF Publikationen ein blätterbares Flipbook mit unserer einzigartigen Google optimierten e-Paper Software.
ERGEBNISSE 97<br />
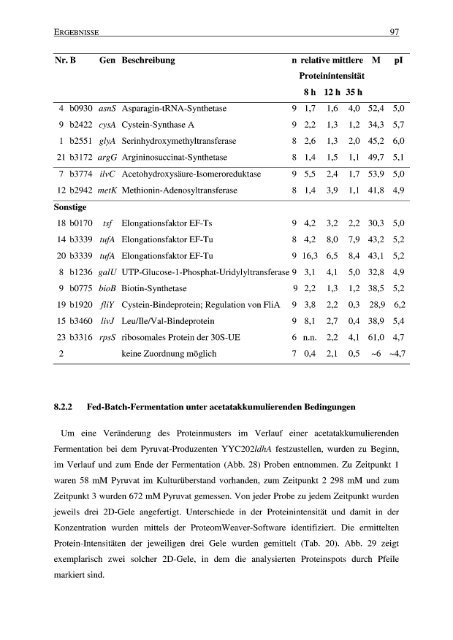
Nr. B Gen Beschreibung<br />
4 b0930 asnS Asparagin-tRNA-Synthetase<br />
9 b2422 cysA Cystein-Synthase A<br />
1 b2551 glyA Serinhydroxymethyltransferase<br />
21 b3172 argG Argininosuccinat-Synthetase<br />
7 b3774 ilvC Acetohydroxysäure-Isomeroreduktase<br />
12 b2942 metK Methionin-Adenosyltransferase<br />
Sonstige<br />
n relative mittlere M<br />
Proteinintensität<br />
9 1,7<br />
9 2,2<br />
8 2,6<br />
8 1,4<br />
8h 12h 35h<br />
18 b0170 tsf Elongationsfaktor EF-Ts 9 4,2 3,2<br />
14 b3339 tufA Elongationsfaktor EF-Tu 8 4,2 8,0<br />
20 b3339 tufA Elongationsfaktor EF-Tu 9 16,3 6,5<br />
8 b1236 galU UTP-Glucose-l-Phosphat-Uridylyltransferase 9 3,1 4,1<br />
9 b0775 bioB Biotin-Synthetase 9 2,2 1,3<br />
19 b1920 fiY Cystein-Bindeprotein ; Regulation von F1iA 9 3,8 2,2<br />
15 b3460 livj Leu/Ile/Val-Bindeprotein 9 8,1 2,7<br />
23 b3316 rpsS ribosomales Protein der 30S-UE 6 n .n . 2,2<br />
2 keine Zuordnung möglich 7 0,4 2,1<br />
pl<br />
1,6 4,0 52,4 5,0<br />
1,3 1,2 34,3 5,7<br />
1,3 2,0 45,2 6,0<br />
1,5 1,1 49,7 5,1<br />
9 5,5 2,4 1,7 53,9 5,0<br />
8 1,4 3,9 1,1 41,8 4,9<br />
8 .2 .2 Fed-Batch-Fermentation unter acetatakkumulierenden Bedingungen<br />
2,2 30,3 5,0<br />
7,9 43,2 5,2<br />
8,4 43,1 5,2<br />
5,0 32,8 4,9<br />
1,2 38,5 5,2<br />
0,3 28,9 6,2<br />
0,4 38,9 5,4<br />
4,1 61,0 4,7<br />
0,5 -6 -4,7<br />
Um eine Veränderung des Proteinmusters im Verlauf einer acetatakkumulierenden<br />
Fermentation bei dem <strong>Pyruvat</strong>-Produzenten YYC202ldhA festzustellen, wurden zu Beginn,<br />
im Verlauf und zum Ende der Fermentation (Abb . 28) Proben entnommen . Zu Zeitpunkt 1<br />
waren 58 mM <strong>Pyruvat</strong> im Kulturüberstand vorhanden, zum Zeitpunkt 2 298 mM und zum<br />
Zeitpunkt 3 wurden 672 mM <strong>Pyruvat</strong> gemessen . Von jeder Probe zu jedem Zeitpunkt wurden<br />
jeweils drei 2D-Gele angefertigt . Unterschiede in der Proteinintensität und damit in der<br />
Konzentration wurden mittels der ProteomWeaver-Software identifiziert . Die ermittelten<br />
Protein-Intensitäten der jeweiligen drei Gele wurden gemittelt (Tab . 20) . Abb . 29 zeigt<br />
exemplarisch zwei solcher 2D-Gele, in dem die analysierten Proteinspots <strong>durch</strong> Pfeile<br />
markiert sind .