Pyruvat-Produktion durch acetatauxotrophe - JUWEL ...
Pyruvat-Produktion durch acetatauxotrophe - JUWEL ...
Pyruvat-Produktion durch acetatauxotrophe - JUWEL ...
Sie wollen auch ein ePaper? Erhöhen Sie die Reichweite Ihrer Titel.
YUMPU macht aus Druck-PDFs automatisch weboptimierte ePaper, die Google liebt.
ERGEBNISSE<br />
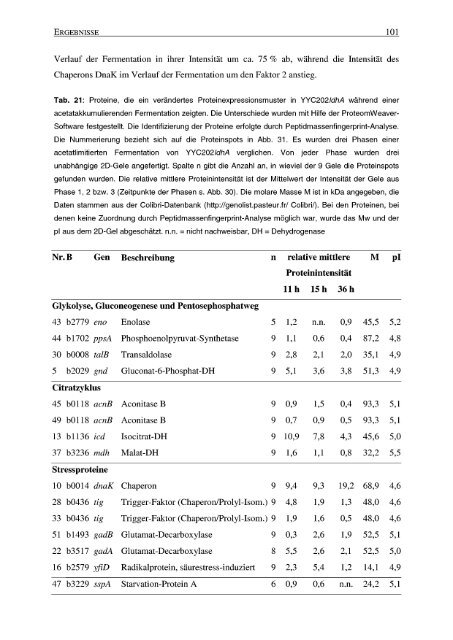
Verlauf der Fermentation in ihrer Intensität um ca . 75 % ab, während die Intensität des<br />
Chaperons DnaK im Verlauf der Fermentation um den Faktor 2 anstieg .<br />
Tab . 21 : Proteine, die ein verändertes Proteinexpressionsmuster in YYC2021dhA während einer<br />
acetatakkumulierenden Fermentation zeigten. Die Unterschiede wurden mit Hilfe der ProteomWeaver-<br />
Software festgestellt . Die Identifizierung der Proteine erfolgte <strong>durch</strong> Peptidmassenfingerprint-Analyse .<br />
Die Nummerierung bezieht sich auf die Proteinspots in Abb . 31 . Es wurden drei Phasen einer<br />
acetatlimitierten Fermentation von YYC2021dhA verglichen . Von jeder Phase wurden drei<br />
unabhängige 2D-Gele angefertigt . Spalte n gibt die Anzahl an, in wieviel der 9 Gele die Proteinspots<br />
gefunden wurden . Die relative mittlere Proteinintensität ist der Mittelwert der Intensität der Gele aus<br />
Phase 1, 2 bzw . 3 (Zeitpunkte der Phasen s . Abb . 30) . Die molare Masse M ist in kDa angegeben, die<br />
Daten stammen aus der Colibri-Datenbank (http ://genolist.pasteur .fr/ Colibri~ . Bei den Proteinen, bei<br />
denen keine Zuordnung <strong>durch</strong> Peptidmassenfingerprint-Analyse möglich war, wurde das Mw und der<br />
pl aus dem 2D-Gel abgeschätzt . n .n . = nicht nachweisbar, DH = Dehydrogenase<br />
Nr. B Gen Beschreibung<br />
Glykolyse, Gluconeogenese und Pentosephosphatweg<br />
43 b2779 eno Enolase<br />
44 b1702 ppsA Phosphoenolpyruvat-Synthetase<br />
30 b0008 talB Transaldolase<br />
5 b2029 gnd Gluconat-6-Phosphat-DH<br />
Citratzyklus<br />
45 b0118 acnB<br />
49 b0118 acnB<br />
13 b1136 icd<br />
37 b3236 mdh<br />
Stressproteine<br />
10 b0014 dnaK<br />
28 b0436 tig<br />
33 b0436 tig<br />
51 b1493 gadB<br />
22 b3517 gadA<br />
16 b2579 yfiD<br />
Aconitase B<br />
Aconitase B<br />
Isocitrat-DH<br />
Malat-DH<br />
Chaperon 9<br />
Trigger-Faktor (Chaperon/Prolyl-Isom .) 9<br />
Trigger-Faktor (Chaperon/Prolyl-Isom .) 9<br />
Glutamat-Decarboxylase 9<br />
Glutamat-Decarboxylase 8<br />
Radikalprotein, säurestress-induziert 9<br />
47 b3229 sspA Starvation-Protein A<br />
n<br />
5<br />
9<br />
9<br />
9<br />
relative mittlere M pl<br />
Proteinintensität<br />
llh 15h 36h<br />
6 0,9<br />
1,2 n.n . 0,9 45,5 5,2<br />
1,1 0,6 0,4 87,2 4,8<br />
2,8 2,1 2,0 35,1 4,9<br />
5,1 3,6 3,8 51,3 4,9<br />
9 0,9 1,5 0,4 93,3 5,1<br />
9 0,7 0,9 0,5 93,3 5,1<br />
9 10,9 7,8 4,3 45,6 5,0<br />
9 1,6 1,1 0,8 32,2 5,5<br />
9,4 9,3 19,2 68,9 4,6<br />
4,8 1,9 1,3 48,0 4,6<br />
1,9 1,6 0,5 48,0 4,6<br />
0,3 2,6 1,9 52,5 5,1<br />
5,5 2,6 2,1 52,5 5,0<br />
2,3 5,4 1,2 14,1 4,9<br />
0,6 n.n . 24,2 5,1