Pyruvat-Produktion durch acetatauxotrophe - JUWEL ...
Pyruvat-Produktion durch acetatauxotrophe - JUWEL ...
Pyruvat-Produktion durch acetatauxotrophe - JUWEL ...
Sie wollen auch ein ePaper? Erhöhen Sie die Reichweite Ihrer Titel.
YUMPU macht aus Druck-PDFs automatisch weboptimierte ePaper, die Google liebt.
ERGEBNISSE 79<br />
" Region I<br />
Gen mRNA- p<br />
Level<br />
Region II<br />
Gen mRNA- p<br />
7 .2 Vergleich des Transkriptoms von <strong>Pyruvat</strong>-Produzent (YYC202) und Wildtyp<br />
(MG1655) bei Wachstum in Glucose-Minimalmedium mit Acetat<br />
In einer zweiten Serie von Transkriptomanalysen mit DNA-Chips wurde der Stamm<br />
YYC202 mit dem Wildtyp-Stamm MG1655 verglichen . Dabei sollten sowohl<br />
pyruvatspezifische als auch stamm-spezifische Unterschiede detektiert werden . Die Stämme<br />
wurden in M9-Minimalmedium mit 10 mM Glucose und 2 mM Acetat kultiviert . Unter<br />
diesen Bedingungen produziert YYC202 <strong>Pyruvat</strong>, nicht aber MG1655 . Die Kultivierung<br />
erfolgte über vier Tage (-200 Generationen) <strong>durch</strong> fünfmaliges Überimpfen, um eine<br />
Adaptation an das entsprechende Medium zu gewährleisten . Die Hauptkultur zur Isolierung<br />
von RNA wurde in der exponentiellen Phase bei einer OD600 zwischen 0,2 und 0,4 geerntet .<br />
Die Kulturen von YYC202 und MG1655 hatten beide eine <strong>durch</strong>schnittliche Wachstumsrate<br />
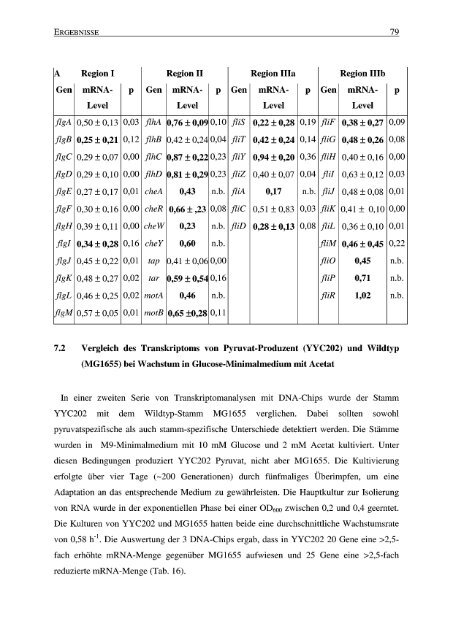
von 0,58 h-1 . Die Auswertung der 3 DNA-Chips ergab, dass in YYC202 20 Gene eine >2,5-<br />
fach erhöhte mRNA-Menge gegenüber MG1655 aufwiesen und 25 Gene eine >2,5-fach<br />
reduzierte mRNA-Menge (Tab . 16) .<br />
Level<br />
Region IIIa<br />
Gen mRNA- p<br />
Level<br />
Region IIIb<br />
Gen mRNA- p<br />
Level<br />
flgA 0,50 ± 0,13 0,03 flhA 0,76 ± 0,09 0,10 flis 0,22 ± 0,28 0,19 fliF 0,38 ± 0,27 0,09<br />
flgB 0,25 ± 0,21 0,12 flhB 0,42 ± 0,24 0,04 fliT 0,42 ± 0,24 0,14 fliG 0,48 ± 0,26 0,08<br />
flgc 0,29 ± 0,07 0,00 flhC 0,87 ± 0,22 0,23 fliy 0,94 ± 0,20 0,36 fliH 0,40 ± 0,16 0,00<br />
flgD 0,29 ± 0,10 0,00 flhD 0,81 ± 0,29 0,23 fliz 0,40 ± 0,07 0,04 fliI 0,63 ± 0,12 0,03<br />
flgE 0,27 ± 0,17 0,01 cheA 0,43 n .b . fliA 0,17 n .b . flij 0,48 ± 0,08 0,01<br />
flgF 0,30 ± 0,16 0,00 cheR 0,66 ±,23 0,08 flic 0,51 ± 0,83 0,03 fliK 0,41 ± 0,10 0,00<br />
flgH 0,39 ± 0,11 0,00 cheW 0,23 n .b . fliD 0,28 ± 0,13 0,08 fliL 0,36 ± 0,10 0,01<br />
flgl 0,34 ± 0,28 0,16 cheY 0,60 n .b . flim 0,46 ± 0,45 0,22<br />
flgj 0,45 ± 0,22 0,01 tap 0,41 ± 0,06 0,00 fli0 0,45 n .b .<br />
flgK 0,48 ± 0,27 0,02 tar 0,59 ± 0,54 0,16 flip 0,71 n .b .<br />
flgL 0,46 ± 0,25 0,02 motA 0,46 n .b . fliR 1,02 n .b .<br />
gM 0,57 ± 0,05 0,01 motB 0,65 ±0,28 0,11