RCGI V31 N63
You also want an ePaper? Increase the reach of your titles
YUMPU automatically turns print PDFs into web optimized ePapers that Google loves.
ORBE SOSA, Z., MARTINEZ NÚÑEZ, M.A. Y VILLALOBOS LÓPEZ, M.A.<br />
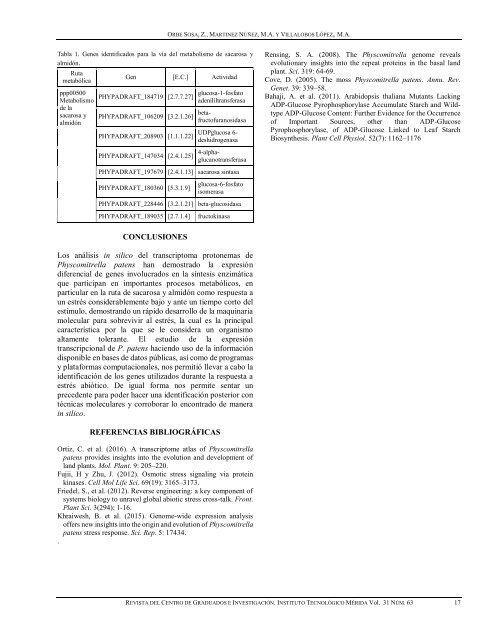
Tabla 1. Genes identificados para la vía del metabolismo de sacarosa y<br />
almidón.<br />
Ruta<br />
metabólica<br />
ppp00500<br />
Metabolismo<br />
de la<br />
sacarosa y<br />
almidón<br />
Gen [E.C.] Actividad<br />
PHYPADRAFT_184719 [2.7.7.27] glucosa-1-fosfato<br />
adenililtransferasa<br />
PHYPADRAFT_106209 [3.2.1.26] betafructofuranosidasa<br />
PHYPADRAFT_208903 [1.1.1.22]<br />
UDPglucosa 6-<br />
deshidrogenasa<br />
PHYPADRAFT_147034 [2.4.1.25] 4-alphaglucanotransferasa<br />
PHYPADRAFT_197679 [2.4.1.13] sacarosa sintasa<br />
PHYPADRAFT_180360 [5.3.1.9]<br />
glucosa-6-fosfato<br />
isomerasa<br />
PHYPADRAFT_228446 [3.2.1.21] beta-glucosidasa<br />
PHYPADRAFT_189035 [2.7.1.4]<br />
fructokinasa<br />
Rensing, S. A. (2008). The Physcomitrella genome reveals<br />
evolutionary insights into the repeat proteins in the basal land<br />
plant. Sci. 319: 64-69.<br />
Cove, D. (2005). The moss Physcomitrella patens. Annu. Rev.<br />
Genet. 39: 339–58.<br />
Bahaji, A. et al. (2011). Arabidopsis thaliana Mutants Lacking<br />
ADP-Glucose Pyrophosphorylase Accumulate Starch and Wildtype<br />
ADP-Glucose Content: Further Evidence for the Occurrence<br />
of Important Sources, other than ADP-Glucose<br />
Pyrophosphorylase, of ADP-Glucose Linked to Leaf Starch<br />
Biosynthesis. Plant Cell Physiol. 52(7): 1162–1176<br />
CONCLUSIONES<br />
Los análisis in silico del transcriptoma protonemas de<br />
Physcomitrella patens han demostrado la expresión<br />
diferencial de genes involucrados en la síntesis enzimática<br />
que participan en importantes procesos metabólicos, en<br />
particular en la ruta de sacarosa y almidón como respuesta a<br />
un estrés considerablemente bajo y ante un tiempo corto del<br />
estímulo, demostrando un rápido desarrollo de la maquinaria<br />
molecular para sobrevivir al estrés, la cual es la principal<br />
característica por la que se le considera un organismo<br />
altamente tolerante. El estudio de la expresión<br />
transcripcional de P. patens haciendo uso de la información<br />
disponible en bases de datos públicas, así como de programas<br />
y plataformas computacionales, nos permitió llevar a cabo la<br />
identificación de los genes utilizados durante la respuesta a<br />
estrés abiótico. De igual forma nos permite sentar un<br />
precedente para poder hacer una identificación posterior con<br />
técnicas moleculares y corroborar lo encontrado de manera<br />
in silico.<br />
REFERENCIAS BIBLIOGRÁFICAS<br />
Ortiz, C. et al. (2016). A transcriptome atlas of Physcomitrella<br />
patens provides insights into the evolution and development of<br />
land plants. Mol. Plant. 9: 205–220.<br />
Fujii, H y Zhu, J. (2012). Osmotic stress signaling via protein<br />
kinases. Cell Mol Life Sci. 69(19): 3165–3173.<br />
Friedel, S., et al. (2012). Reverse engineering: a key component of<br />
systems biology to unravel global abiotic stress cross-talk. Front.<br />
Plant Sci. 3(294): 1-16.<br />
Khraiwesh, B. et al. (2015). Genome-wide expression analysis<br />
offers new insights into the origin and evolution of Physcomitrella<br />
patens stress response. Sci. Rep. 5: 17434.<br />
.<br />
REVISTA DEL CENTRO DE GRADUADOS E INVESTIGACIÓN. INSTITUTO TECNOLÓGICO MÉRIDA Vol. 31 NÚM. 63 17