PDF 5.972kB - TOBIAS-lib - Universität Tübingen
PDF 5.972kB - TOBIAS-lib - Universität Tübingen
PDF 5.972kB - TOBIAS-lib - Universität Tübingen
Sie wollen auch ein ePaper? Erhöhen Sie die Reichweite Ihrer Titel.
YUMPU macht aus Druck-PDFs automatisch weboptimierte ePaper, die Google liebt.
ERGEBNISSE<br />
Immunstimulatoren lokalisierten, T7-Promotor enthielten, war eine Überprüfung der<br />
E2-Transkription für diese Konstrukte nicht möglich.<br />
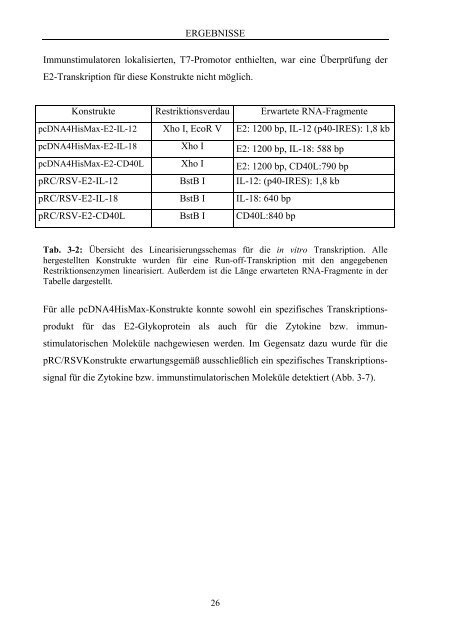
Konstrukte Restriktionsverdau Erwartete RNA-Fragmente<br />
pcDNA4HisMax-E2-IL-12 Xho I, EcoR V E2: 1200 bp, IL-12 (p40-IRES): 1,8 kb<br />
pcDNA4HisMax-E2-IL-18 Xho I E2: 1200 bp, IL-18: 588 bp<br />
pcDNA4HisMax-E2-CD40L Xho I E2: 1200 bp, CD40L:790 bp<br />
pRC/RSV-E2-IL-12 BstB I IL-12: (p40-IRES): 1,8 kb<br />
pRC/RSV-E2-IL-18 BstB I IL-18: 640 bp<br />
pRC/RSV-E2-CD40L BstB I CD40L:840 bp<br />
Tab. 3-2: Übersicht des Linearisierungsschemas für die in vitro Transkription. Alle<br />
hergestellten Konstrukte wurden für eine Run-off-Transkription mit den angegebenen<br />
Restriktionsenzymen linearisiert. Außerdem ist die Länge erwarteten RNA-Fragmente in der<br />
Tabelle dargestellt.<br />
Für alle pcDNA4HisMax-Konstrukte konnte sowohl ein spezifisches Transkriptions-<br />
produkt für das E2-Glykoprotein als auch für die Zytokine bzw. immun-<br />
stimulatorischen Moleküle nachgewiesen werden. Im Gegensatz dazu wurde für die<br />
pRC/RSVKonstrukte erwartungsgemäß ausschließlich ein spezifisches Transkriptions-<br />
signal für die Zytokine bzw. immunstimulatorischen Moleküle detektiert (Abb. 3-7).<br />
26