Alberto Risueño Pérez - Gredos - Universidad de Salamanca
Alberto Risueño Pérez - Gredos - Universidad de Salamanca
Alberto Risueño Pérez - Gredos - Universidad de Salamanca
You also want an ePaper? Increase the reach of your titles
YUMPU automatically turns print PDFs into web optimized ePapers that Google loves.
Tesis Doctoral<br />
En un organismo pluricelular la división celular está ligada a la diferenciación, proceso que se<br />
controla mediante un programa <strong>de</strong> crecimiento y cambio celular necesario para la<br />
especialización. Alteraciones externas pue<strong>de</strong>n inhibir esta diferenciación y especialización,<br />
manteniendo la célula en un estado <strong>de</strong> inmadurez quiescente con una alta capacidad <strong>de</strong><br />
división (von Wangenheim and Peterson, 2008). Esto significa que las enfermeda<strong>de</strong>s<br />
neoplásicas tumorales implican un bloqueo en el proceso <strong>de</strong> diferenciación, que <strong>de</strong>bería<br />
suponer una limitación en la expresión <strong>de</strong> genes específicos <strong>de</strong> tejido o <strong>de</strong> tipo celular lo cual<br />
es precisamente lo que <strong>de</strong>scubrimos en nuestros datos <strong>de</strong> la figura 4.8 y 4.9. A<strong>de</strong>más, la<br />
comparación entre S3 vs S1B <strong>de</strong> la figura 4.9 sugiere que no solo existe bloqueo en la<br />
diferenciación celular durante el proceso <strong>de</strong> carcinogénesis, sino que también existen ciertas<br />
diferencias evolutivas entre los genes sobre e infra-‐expresados en la progresión tumoral. De<br />
este resultado, y con los datos <strong>de</strong> conservación <strong>de</strong> los HKG y TSG, se <strong>de</strong>spren<strong>de</strong> que los genes<br />
sobre-‐expresados en cáncer son genes altamente conservados constitutivos, asociados a<br />
activida<strong>de</strong>s esenciales <strong>de</strong> la célula –como crecimiento y replicación–, y que realizan funciones<br />
importantes para su mantenimiento como los procesos energéticos, síntesis <strong>de</strong> proteínas, ciclo<br />
celular, etc. Por contra, los genes reprimidos son genes propios <strong>de</strong> sistemas pluricelulares<br />
especializados, como genes específicos <strong>de</strong> tejido, que son innecesarios si el proceso <strong>de</strong><br />
diferenciación se bloquea y que no están presentes en organismos unicelulares más sencillos y<br />
arcaicos.<br />
Finalmente, para comprobar si la variación entre la conservación <strong>de</strong> los genes reprimidos y<br />
activados en cáncer se correspon<strong>de</strong> con su nivel <strong>de</strong> especificidad en tejidos, se realizó la<br />
intersección <strong>de</strong> los genes específicos <strong>de</strong> tejido i<strong>de</strong>ntificados en este trabajo (786 TSG<br />
<strong>de</strong>tectados a nivel 2, ver apartado 4.2.4) con los genes i<strong>de</strong>ntificados sobre-‐expresados e infra-‐<br />
expresados en cinco tipos <strong>de</strong> cáncer estudiados.<br />
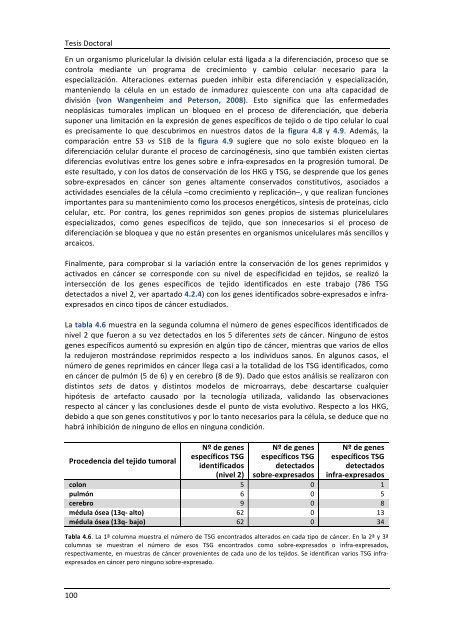
La tabla 4.6 muestra en la segunda columna el número <strong>de</strong> genes específicos i<strong>de</strong>ntificados <strong>de</strong><br />
nivel 2 que fueron a su vez <strong>de</strong>tectados en los 5 diferentes sets <strong>de</strong> cáncer. Ninguno <strong>de</strong> estos<br />
genes específicos aumentó su expresión en algún tipo <strong>de</strong> cáncer, mientras que varios <strong>de</strong> ellos<br />
la redujeron mostrándose reprimidos respecto a los individuos sanos. En algunos casos, el<br />
número <strong>de</strong> genes reprimidos en cáncer llega casi a la totalidad <strong>de</strong> los TSG i<strong>de</strong>ntificados, como<br />
en cáncer <strong>de</strong> pulmón (5 <strong>de</strong> 6) y en cerebro (8 <strong>de</strong> 9). Dado que estos análisis se realizaron con<br />
distintos sets <strong>de</strong> datos y distintos mo<strong>de</strong>los <strong>de</strong> microarrays, <strong>de</strong>be <strong>de</strong>scartarse cualquier<br />
hipótesis <strong>de</strong> artefacto causado por la tecnología utilizada, validando las observaciones<br />
respecto al cáncer y las conclusiones <strong>de</strong>s<strong>de</strong> el punto <strong>de</strong> vista evolutivo. Respecto a los HKG,<br />
<strong>de</strong>bido a que son genes constitutivos y por lo tanto necesarios para la célula, se <strong>de</strong>duce que no<br />
habrá inhibición <strong>de</strong> ninguno <strong>de</strong> ellos en ninguna condición.<br />
Proce<strong>de</strong>ncia <strong>de</strong>l tejido tumoral<br />
100<br />
Nº <strong>de</strong> genes<br />
específicos TSG<br />
i<strong>de</strong>ntificados<br />
(nivel 2)<br />
Nº <strong>de</strong> genes<br />
específicos TSG<br />
<strong>de</strong>tectados<br />
sobre-‐expresados<br />
Nº <strong>de</strong> genes<br />
específicos TSG<br />
<strong>de</strong>tectados<br />
infra-‐expresados<br />
colon 5 0 1<br />
pulmón 6 0 5<br />
cerebro 9 0 8<br />
médula ósea (13q-‐ alto) 62 0 13<br />
médula ósea (13q-‐ bajo) 62 0 34<br />
Tabla 4.6. La 1ª columna muestra el número <strong>de</strong> TSG encontrados alterados en cada tipo <strong>de</strong> cáncer. En la 2ª y 3ª<br />
columnas se muestran el número <strong>de</strong> esos TSG encontrados como sobre-‐expresados o infra-‐expresados,<br />
respectivamente, en muestras <strong>de</strong> cáncer provenientes <strong>de</strong> cada uno <strong>de</strong> los tejidos. Se i<strong>de</strong>ntifican varios TSG infra-‐<br />
expresados en cáncer pero ninguno sobre-‐expresado.