Alberto Risueño Pérez - Gredos - Universidad de Salamanca
Alberto Risueño Pérez - Gredos - Universidad de Salamanca
Alberto Risueño Pérez - Gredos - Universidad de Salamanca
You also want an ePaper? Increase the reach of your titles
YUMPU automatically turns print PDFs into web optimized ePapers that Google loves.
Tesis Doctoral<br />
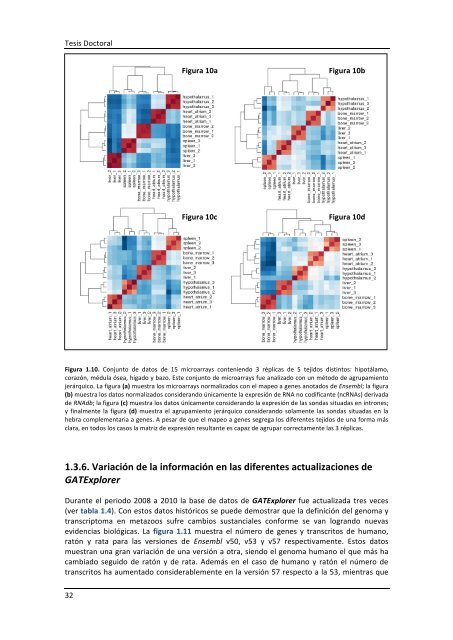
Figura 1.10. Conjunto <strong>de</strong> datos <strong>de</strong> 15 microarrays conteniendo 3 réplicas <strong>de</strong> 5 tejidos distintos: hipotálamo,<br />
corazón, médula ósea, hígado y bazo. Este conjunto <strong>de</strong> microarrays fue analizado con un método <strong>de</strong> agrupamiento<br />
jerárquico. La figura (a) muestra los microarrays normalizados con el mapeo a genes anotados <strong>de</strong> Ensembl; la figura<br />
(b) muestra los datos normalizados consi<strong>de</strong>rando únicamente la expresión <strong>de</strong> RNA no codificante (ncRNAs) <strong>de</strong>rivada<br />
<strong>de</strong> RNAdb; la figura (c) muestra los datos únicamente consi<strong>de</strong>rando la expresión <strong>de</strong> las sondas situadas en intrones;<br />
y finalmente la figura (d) muestra el agrupamiento jerárquico consi<strong>de</strong>rando solamente las sondas situadas en la<br />
hebra complementaria a genes. A pesar <strong>de</strong> que el mapeo a genes segrega los diferentes tejidos <strong>de</strong> una forma más<br />
clara, en todos los casos la matriz <strong>de</strong> expresión resultante es capaz <strong>de</strong> agrupar correctamente las 3 réplicas.<br />
1.3.6. Variación <strong>de</strong> la información en las diferentes actualizaciones <strong>de</strong><br />
GATExplorer<br />
Durante el periodo 2008 a 2010 la base <strong>de</strong> datos <strong>de</strong> GATExplorer fue actualizada tres veces<br />
(ver tabla 1.4). Con estos datos históricos se pue<strong>de</strong> <strong>de</strong>mostrar que la <strong>de</strong>finición <strong>de</strong>l genoma y<br />
transcriptoma en metazoos sufre cambios sustanciales conforme se van logrando nuevas<br />
evi<strong>de</strong>ncias biológicas. La figura 1.11 muestra el número <strong>de</strong> genes y transcritos <strong>de</strong> humano,<br />
ratón y rata para las versiones <strong>de</strong> Ensembl v50, v53 y v57 respectivamente. Estos datos<br />
muestran una gran variación <strong>de</strong> una versión a otra, siendo el genoma humano el que más ha<br />
cambiado seguido <strong>de</strong> ratón y <strong>de</strong> rata. A<strong>de</strong>más en el caso <strong>de</strong> humano y ratón el número <strong>de</strong><br />
transcritos ha aumentado consi<strong>de</strong>rablemente en la versión 57 respecto a la 53, mientras que<br />
32<br />
Figura 10a Figura 10b<br />
Figura 10c Figura 10d