- Page 1: Bioinformática aplicada a estudios
- Page 5 and 6: Índice INTRODUCCIÓN GENERAL .....
- Page 7 and 8: Introducción general Bioinformáti
- Page 9 and 10: Figura 2. Proceso de transcripción
- Page 11 and 12: Introducción general caciones, las
- Page 13 and 14: Objetivos Introducción general La
- Page 15 and 16: Capítulo 1 1.1.1. Bases de datos d
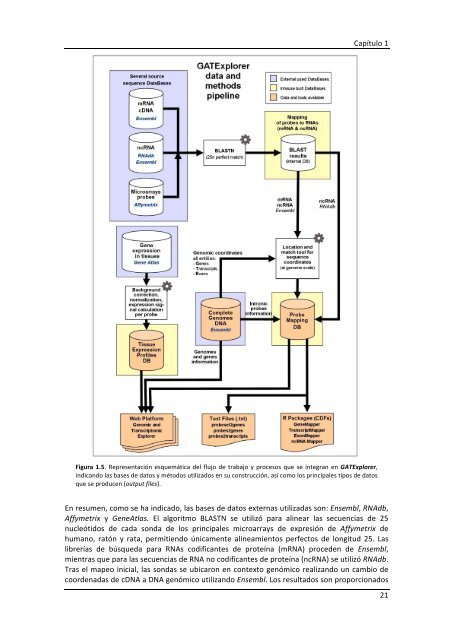
- Page 17 and 18: Capítulo 1 sondas core y su inform
- Page 19 and 20: caaatgacttgctattattgatggc 225 694 c
- Page 21 and 22: presentes en el fichero. Capítulo
- Page 23: Capítulo 1 Mus musculus MG_U74Av2
- Page 27 and 28: Capítulo 1 Paso 2 Descripción: As
- Page 29 and 30: Capítulo 1 A la hora de escribir e
- Page 31 and 32: Capítulo 1 en regiones no codifica
- Page 33 and 34: Capítulo 1 Para optimizar la preci
- Page 35 and 36: Figura 1.9a. Distribución del núm
- Page 37 and 38: Capítulo 1 por contraste el númer
- Page 39 and 40: Capítulo 1 (cromosoma, locus, exon
- Page 41 and 42: Capítulo 1 figura 1.16). Además d
- Page 43 and 44: Capítulo 1 exhaustivo en este ámb
- Page 45 and 46: Capítulo 1 su presentación y deta
- Page 47: Capítulo 1 adaptación para los mi
- Page 50 and 51: Tesis Doctoral pueden agrupar en: t
- Page 52 and 53: Tesis Doctoral enfermedad a través
- Page 54 and 55: Tesis Doctoral los genes encontrado
- Page 56 and 57: Tesis Doctoral real (RT-‐PCR).
- Page 58 and 59: Tesis Doctoral muestras (ver figura
- Page 60 and 61: Tesis Doctoral subtipo fueron: 0.97
- Page 62 and 63: Tesis Doctoral En este trabajo se h
- Page 64 and 65: Tesis Doctoral permitiría, sin dud
- Page 66 and 67: Tesis Doctoral inclusión entre 0 y
- Page 68 and 69: Tesis Doctoral exacto del número d
- Page 70 and 71: Tesis Doctoral Los valores extremos
- Page 72 and 73: Tesis Doctoral dicho, la comparaci
- Page 74 and 75:
Tesis Doctoral 70 Figura 3.6. Los d
- Page 76 and 77:
Tesis Doctoral 3.8.b). Sin embargo
- Page 78 and 79:
Tesis Doctoral Human Exon 1.0. La l
- Page 80 and 81:
Tesis Doctoral 76 Figura 3.10. Curv
- Page 82 and 83:
Tesis Doctoral 78 Figura 3.10 (cont
- Page 84 and 85:
Tesis Doctoral del inicio del ranki
- Page 87 and 88:
Capítulo 4 Análisis de coexpresi
- Page 89 and 90:
Capítulo 4 los genes y la perspect
- Page 91 and 92:
Capítulo 4 Utilizando el set de da
- Page 93 and 94:
ENSG00000142541 RPL13A small nucleo
- Page 95 and 96:
Capítulo 4 Para encontrar los gene
- Page 97 and 98:
Capítulo 4 ENSG00000134287 ARF3 AD
- Page 99 and 100:
Capítulo 4 Figura 4.3. Red de coex
- Page 101 and 102:
Capítulo 4 Si analizamos los genes
- Page 103 and 104:
Capítulo 4 se hizo comparando cont
- Page 105:
4.4. Discusión y posible trabajo f
- Page 108 and 109:
Tesis Doctoral exones, y diseñando
- Page 110 and 111:
Tesis Doctoral expression and isofo
- Page 112 and 113:
Tesis Doctoral 37, e107. Gardina, P
- Page 114 and 115:
Tesis Doctoral and survival in chro
- Page 116 and 117:
Tesis Doctoral Roth, R.B., Hevezi,
- Page 118 and 119:
Tesis Doctoral Xi, L., Feber, A., G
- Page 121 and 122:
Risueño et al. BMC Bioinformatics
- Page 123 and 124:
Risueño et al. BMC Bioinformatics
- Page 125 and 126:
Risueño et al. BMC Bioinformatics
- Page 127 and 128:
Risueño et al. BMC Bioinformatics
- Page 129 and 130:
Risueño et al. BMC Bioinformatics
- Page 131 and 132:
Risueño et al. BMC Bioinformatics
- Page 133 and 134:
ORIGINAL ARTICLE Deregulation of mi
- Page 135 and 136:
Targets component of miRecords inte
- Page 137 and 138:
log 10 2-ΔCt -2.00 -4.00 -6.00 -8.
- Page 139 and 140:
Table 4 Potential microRNA (miRNA)-
- Page 141 and 142:
myeloma pathogenesis. Proc Natl Aca
- Page 143 and 144:
genetic subtypes of CLL show differ
- Page 145 and 146:
Table 2. Cont. Up-regulated Down-re
- Page 147 and 148:
206 underexpressed in the 13q-H gro
- Page 149 and 150:
Table 3. Most significant target ge
- Page 151 and 152:
Discussion 13q deletion (13q-) is t
- Page 153 and 154:
patients with 17p and 11q deletions
- Page 155 and 156:
Human Gene Coexpression Landscape:
- Page 157 and 158:
The similarity and proximity of the
- Page 159 and 160:
As described in Methods we use a co
- Page 161 and 162:
all data points of coexpression pai
- Page 163 and 164:
Table 1. This work (2008) Pathway N
- Page 165 and 166:
In conclusion, the functional consi
- Page 167 and 168:
a total set of 48 microarrays. The
- Page 169 and 170:
original article Annals of Oncology
- Page 171 and 172:
Annals of Oncology original article
- Page 173 and 174:
Annals of Oncology original article
- Page 175 and 176:
Annals of Oncology original article