Alberto Risueño Pérez - Gredos - Universidad de Salamanca
Alberto Risueño Pérez - Gredos - Universidad de Salamanca
Alberto Risueño Pérez - Gredos - Universidad de Salamanca
Create successful ePaper yourself
Turn your PDF publications into a flip-book with our unique Google optimized e-Paper software.
Capítulo 2<br />
el sistema AutoMACs automated separation system (Milteyi-‐Biotec, Auburn CA, USA) elevando<br />
la pureza por encima <strong>de</strong>l 90%. La clasificación <strong>de</strong> los pacientes se hizo en función <strong>de</strong> las<br />
aberraciones cromosómicas, obteniendo seis subtipos <strong>de</strong> MM que representan las alteraciones<br />
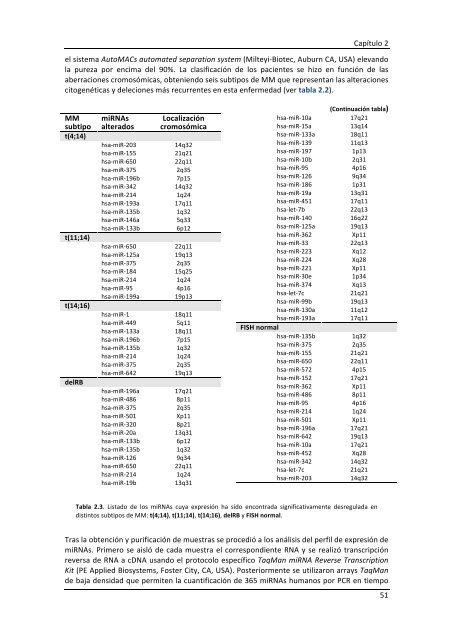
citogenéticas y <strong>de</strong>leciones más recurrentes en esta enfermedad (ver tabla 2.2).<br />
MM<br />
subtipo<br />
t(4;14)<br />
t(11;14)<br />
t(14;16)<br />
<strong>de</strong>lRB<br />
miRNAs<br />
alterados<br />
Localización<br />
cromosómica<br />
hsa-‐miR-‐203 14q32<br />
hsa-‐miR-‐155 21q21<br />
hsa-‐miR-‐650 22q11<br />
hsa-‐miR-‐375 2q35<br />
hsa-‐miR-‐196b 7p15<br />
hsa-‐miR-‐342 14q32<br />
hsa-‐miR-‐214 1q24<br />
hsa-‐miR-‐193a 17q11<br />
hsa-‐miR-‐135b 1q32<br />
hsa-‐miR-‐146a 5q33<br />
hsa-‐miR-‐133b 6p12<br />
hsa-‐miR-‐650 22q11<br />
hsa-‐miR-‐125a 19q13<br />
hsa-‐miR-‐375 2q35<br />
hsa-‐miR-‐184 15q25<br />
hsa-‐miR-‐214 1q24<br />
hsa-‐miR-‐95 4p16<br />
hsa-‐miR-‐199a 19p13<br />
hsa-‐miR-‐1 18q11<br />
hsa-‐miR-‐449 5q11<br />
hsa-‐miR-‐133a 18q11<br />
hsa-‐miR-‐196b 7p15<br />
hsa-‐miR-‐135b 1q32<br />
hsa-‐miR-‐214 1q24<br />
hsa-‐miR-‐375 2q35<br />
hsa-‐miR-‐642 19q13<br />
hsa-‐miR-‐196a 17q21<br />
hsa-‐miR-‐486 8p11<br />
hsa-‐miR-‐375 2q35<br />
hsa-‐miR-‐501 Xp11<br />
hsa-‐miR-‐320 8p21<br />
hsa-‐miR-‐20a 13q31<br />
hsa-‐miR-‐133b 6p12<br />
hsa-‐miR-‐135b 1q32<br />
hsa-‐miR-‐126 9q34<br />
hsa-‐miR-‐650 22q11<br />
hsa-‐miR-‐214 1q24<br />
hsa-‐miR-‐19b 13q31<br />
hsa-‐miR-‐10a<br />
(Continuación tabla)<br />
17q21<br />
hsa-‐miR-‐15a 13q14<br />
hsa-‐miR-‐133a 18q11<br />
hsa-‐miR-‐139 11q13<br />
hsa-‐miR-‐197 1p13<br />
hsa-‐miR-‐10b 2q31<br />
hsa-‐miR-‐95 4p16<br />
hsa-‐miR-‐126 9q34<br />
hsa-‐miR-‐186 1p31<br />
hsa-‐miR-‐19a 13q31<br />
hsa-‐miR-‐451 17q11<br />
hsa-‐let-‐7b 22q13<br />
hsa-‐miR-‐140 16q22<br />
hsa-‐miR-‐125a 19q13<br />
hsa-‐miR-‐362 Xp11<br />
hsa-‐miR-‐33 22q13<br />
hsa-‐miR-‐223 Xq12<br />
hsa-‐miR-‐224 Xq28<br />
hsa-‐miR-‐221 Xp11<br />
hsa-‐miR-‐30e 1p34<br />
hsa-‐miR-‐374 Xq13<br />
hsa-‐let-‐7c 21q21<br />
hsa-‐miR-‐99b 19q13<br />
hsa-‐miR-‐130a 11q12<br />
hsa-‐miR-‐193a<br />
FISH normal<br />
17q11<br />
hsa-‐miR-‐135b 1q32<br />
hsa-‐miR-‐375 2q35<br />
hsa-‐miR-‐155 21q21<br />
hsa-‐miR-‐650 22q11<br />
hsa-‐miR-‐572 4p15<br />
hsa-‐miR-‐152 17q21<br />
hsa-‐miR-‐362 Xp11<br />
hsa-‐miR-‐486 8p11<br />
hsa-‐miR-‐95 4p16<br />
hsa-‐miR-‐214 1q24<br />
hsa-‐miR-‐501 Xp11<br />
hsa-‐miR-‐196a 17q21<br />
hsa-‐miR-‐642 19q13<br />
hsa-‐miR-‐10a 17q21<br />
hsa-‐miR-‐452 Xq28<br />
hsa-‐miR-‐342 14q32<br />
hsa-‐let-‐7c 21q21<br />
hsa-‐miR-‐203 14q32<br />
Tabla 2.3. Listado <strong>de</strong> los miRNAs cuya expresión ha sido encontrada significativamente <strong>de</strong>sregulada en<br />
distintos subtipos <strong>de</strong> MM: t(4;14), t(11;14), t(14;16), <strong>de</strong>lRB y FISH normal.<br />
Tras la obtención y purificación <strong>de</strong> muestras se procedió a los análisis <strong>de</strong>l perfil <strong>de</strong> expresión <strong>de</strong><br />
miRNAs. Primero se aisló <strong>de</strong> cada muestra el correspondiente RNA y se realizó transcripción<br />
reversa <strong>de</strong> RNA a cDNA usando el protocolo específico TaqMan miRNA Reverse Transcription<br />
Kit (PE Applied Biosystems, Foster City, CA, USA). Posteriormente se utilizaron arrays TaqMan<br />
<strong>de</strong> baja <strong>de</strong>nsidad que permiten la cuantificación <strong>de</strong> 365 miRNAs humanos por PCR en tiempo<br />
51