Alberto Risueño Pérez - Gredos - Universidad de Salamanca
Alberto Risueño Pérez - Gredos - Universidad de Salamanca
Alberto Risueño Pérez - Gredos - Universidad de Salamanca
You also want an ePaper? Increase the reach of your titles
YUMPU automatically turns print PDFs into web optimized ePapers that Google loves.
Capítulo 2<br />
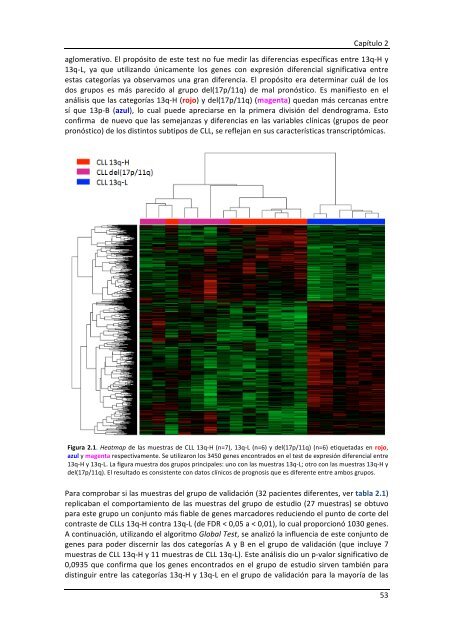
aglomerativo. El propósito <strong>de</strong> este test no fue medir las diferencias específicas entre 13q-‐H y<br />
13q-‐L, ya que utilizando únicamente los genes con expresión diferencial significativa entre<br />
estas categorías ya observamos una gran diferencia. El propósito era <strong>de</strong>terminar cuál <strong>de</strong> los<br />
dos grupos es más parecido al grupo <strong>de</strong>l(17p/11q) <strong>de</strong> mal pronóstico. Es manifiesto en el<br />
análisis que las categorías 13q-‐H (rojo) y <strong>de</strong>l(17p/11q) (magenta) quedan más cercanas entre<br />
sí que 13p-‐B (azul), lo cual pue<strong>de</strong> apreciarse en la primera división <strong>de</strong>l <strong>de</strong>ndrograma. Esto<br />
confirma <strong>de</strong> nuevo que las semejanzas y diferencias en las variables clínicas (grupos <strong>de</strong> peor<br />
pronóstico) <strong>de</strong> los distintos subtipos <strong>de</strong> CLL, se reflejan en sus características transcriptómicas.<br />
Figura 2.1. Heatmap <strong>de</strong> las muestras <strong>de</strong> CLL 13q-‐H (n=7), 13q-‐L (n=6) y <strong>de</strong>l(17p/11q) (n=6) etiquetadas en rojo,<br />
azul y magenta respectivamente. Se utilizaron los 3450 genes encontrados en el test <strong>de</strong> expresión diferencial entre<br />
13q-‐H y 13q-‐L. La figura muestra dos grupos principales: uno con las muestras 13q-‐L; otro con las muestras 13q-‐H y<br />
<strong>de</strong>l(17p/11q). El resultado es consistente con datos clínicos <strong>de</strong> prognosis que es diferente entre ambos grupos.<br />
Para comprobar si las muestras <strong>de</strong>l grupo <strong>de</strong> validación (32 pacientes diferentes, ver tabla 2.1)<br />
replicaban el comportamiento <strong>de</strong> las muestras <strong>de</strong>l grupo <strong>de</strong> estudio (27 muestras) se obtuvo<br />
para este grupo un conjunto más fiable <strong>de</strong> genes marcadores reduciendo el punto <strong>de</strong> corte <strong>de</strong>l<br />
contraste <strong>de</strong> CLLs 13q-‐H contra 13q-‐L (<strong>de</strong> FDR < 0,05 a < 0,01), lo cual proporcionó 1030 genes.<br />
A continuación, utilizando el algoritmo Global Test, se analizó la influencia <strong>de</strong> este conjunto <strong>de</strong><br />
genes para po<strong>de</strong>r discernir las dos categorías A y B en el grupo <strong>de</strong> validación (que incluye 7<br />
muestras <strong>de</strong> CLL 13q-‐H y 11 muestras <strong>de</strong> CLL 13q-‐L). Este análisis dio un p-‐valor significativo <strong>de</strong><br />
0,0935 que confirma que los genes encontrados en el grupo <strong>de</strong> estudio sirven también para<br />
distinguir entre las categorías 13q-‐H y 13q-‐L en el grupo <strong>de</strong> validación para la mayoría <strong>de</strong> las<br />
53