Alberto Risueño Pérez - Gredos - Universidad de Salamanca
Alberto Risueño Pérez - Gredos - Universidad de Salamanca
Alberto Risueño Pérez - Gredos - Universidad de Salamanca
Create successful ePaper yourself
Turn your PDF publications into a flip-book with our unique Google optimized e-Paper software.
Tesis Doctoral<br />
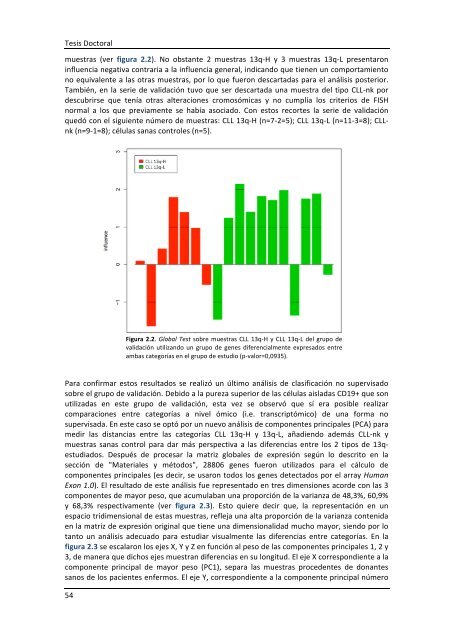
muestras (ver figura 2.2). No obstante 2 muestras 13q-‐H y 3 muestras 13q-‐L presentaron<br />
influencia negativa contraria a la influencia general, indicando que tienen un comportamiento<br />
no equivalente a las otras muestras, por lo que fueron <strong>de</strong>scartadas para el análisis posterior.<br />
También, en la serie <strong>de</strong> validación tuvo que ser <strong>de</strong>scartada una muestra <strong>de</strong>l tipo CLL-‐nk por<br />
<strong>de</strong>scubrirse que tenía otras alteraciones cromosómicas y no cumplía los criterios <strong>de</strong> FISH<br />
normal a los que previamente se había asociado. Con estos recortes la serie <strong>de</strong> validación<br />
quedó con el siguiente número <strong>de</strong> muestras: CLL 13q-‐H (n=7-‐2=5); CLL 13q-‐L (n=11-‐3=8); CLL-‐<br />
nk (n=9-‐1=8); células sanas controles (n=5).<br />
Para confirmar estos resultados se realizó un último análisis <strong>de</strong> clasificación no supervisado<br />
sobre el grupo <strong>de</strong> validación. Debido a la pureza superior <strong>de</strong> las células aisladas CD19+ que son<br />
utilizadas en este grupo <strong>de</strong> validación, esta vez se observó que sí era posible realizar<br />
comparaciones entre categorías a nivel ómico (i.e. transcriptómico) <strong>de</strong> una forma no<br />
supervisada. En este caso se optó por un nuevo análisis <strong>de</strong> componentes principales (PCA) para<br />
medir las distancias entre las categorías CLL 13q-‐H y 13q-‐L, añadiendo a<strong>de</strong>más CLL-‐nk y<br />
muestras sanas control para dar más perspectiva a las diferencias entre los 2 tipos <strong>de</strong> 13q-‐<br />
estudiados. Después <strong>de</strong> procesar la matriz globales <strong>de</strong> expresión según lo <strong>de</strong>scrito en la<br />
sección <strong>de</strong> "Materiales y métodos", 28806 genes fueron utilizados para el cálculo <strong>de</strong><br />
componentes principales (es <strong>de</strong>cir, se usaron todos los genes <strong>de</strong>tectados por el array Human<br />
Exon 1.0). El resultado <strong>de</strong> este análisis fue representado en tres dimensiones acor<strong>de</strong> con las 3<br />
componentes <strong>de</strong> mayor peso, que acumulaban una proporción <strong>de</strong> la varianza <strong>de</strong> 48,3%, 60,9%<br />
y 68,3% respectivamente (ver figura 2.3). Esto quiere <strong>de</strong>cir que, la representación en un<br />
espacio tridimensional <strong>de</strong> estas muestras, refleja una alta proporción <strong>de</strong> la varianza contenida<br />
en la matriz <strong>de</strong> expresión original que tiene una dimensionalidad mucho mayor, siendo por lo<br />
tanto un análisis a<strong>de</strong>cuado para estudiar visualmente las diferencias entre categorías. En la<br />
figura 2.3 se escalaron los ejes X, Y y Z en función al peso <strong>de</strong> las componentes principales 1, 2 y<br />
3, <strong>de</strong> manera que dichos ejes muestran diferencias en su longitud. El eje X correspondiente a la<br />
componente principal <strong>de</strong> mayor peso (PC1), separa las muestras proce<strong>de</strong>ntes <strong>de</strong> donantes<br />
sanos <strong>de</strong> los pacientes enfermos. El eje Y, correspondiente a la componente principal número<br />
54<br />
Figura 2.2. Global Test sobre muestras CLL 13q-‐H y CLL 13q-‐L <strong>de</strong>l grupo <strong>de</strong><br />
validación utilizando un grupo <strong>de</strong> genes diferencialmente expresados entre<br />
ambas categorías en el grupo <strong>de</strong> estudio (p-‐valor=0,0935).