Alberto Risueño Pérez - Gredos - Universidad de Salamanca
Alberto Risueño Pérez - Gredos - Universidad de Salamanca
Alberto Risueño Pérez - Gredos - Universidad de Salamanca
Create successful ePaper yourself
Turn your PDF publications into a flip-book with our unique Google optimized e-Paper software.
ESLiMc ESLiMt FIRMA ARH COSIE<br />
BRE-‐CER 0,834 0,854 0,752 0,784 0,640<br />
BRE-‐HEA 0,708 0,625 0,802 0,882 0,681<br />
BRE-‐LIV 0,804 0,833 0,879 0,884 0,775<br />
BRE-‐MUS 0,894 0,872 0,801 0,858 0,647<br />
BRE-‐TES 0,844 0,856 0,756 0,708 0,636<br />
CER-‐HEA 0,737 0,758 0,715 0,711 0,656<br />
CER-‐LIV 0,763 0,785 0,645 0,735 0,705<br />
CER-‐MUS 0,825 0,763 0,767 0,795 0,728<br />
CER-‐TES 0,820 0,840 0,690 0,768 0,647<br />
HEA-‐LIV 0,799 0,897 0,716 0,853 0,588<br />
HEA-‐MUS 0,854 0,829 0,790 0,807 0,656<br />
HEA-‐TES 0,935 0,940 0,901 0,717 0,886<br />
LIV-‐MUS 0,966 NA 0,736 0,676 0,796<br />
LIV-‐TES 0,789 0,809 0,732 0,773 0,650<br />
MUS-‐TES 0,899 0,931 0,751 0,880 0,768<br />
Media 0,832 0,828 0,762 0,789 0,697<br />
Media pon<strong>de</strong>rada 0,823 0,818 0,746 0,782 0,675<br />
Capítulo 3<br />
Tabla 3.4. Comparativa <strong>de</strong> AUCs <strong>de</strong> las ROCs alcanzada por cada par <strong>de</strong> tejidos y método <strong>de</strong> <strong>de</strong>tección <strong>de</strong> splicing<br />
alternativo estudiados. Se han utilizado dos métodos para promediar los 15 contrastes: media simple y media<br />
pon<strong>de</strong>rada por número <strong>de</strong> positivos (genes con splicing validado). Los dos métodos <strong>de</strong>sarrollados en este trabajo<br />
(ESLiMc y ESLiMt) muestran un mejor rendimiento que algoritmos previamente publicados (FIRMA, ARH y COSIE).<br />
ESLiMt presenta un "NA" en la comparación LIV-‐MUS <strong>de</strong>bido a que ninguna sonda mapea en los genes validados<br />
para par <strong>de</strong> tejidos (ver tabla 3.3).<br />
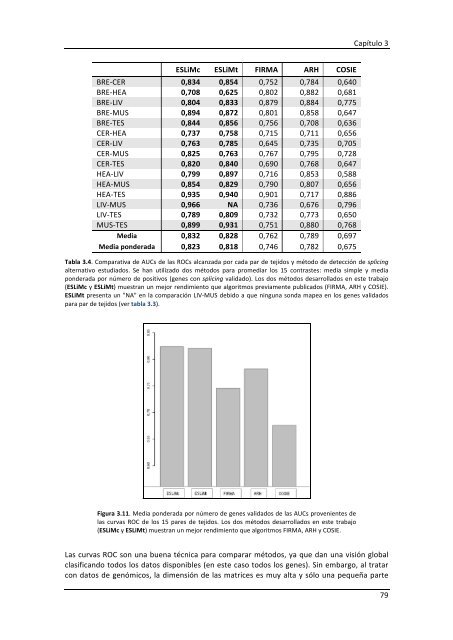
Figura 3.11. Media pon<strong>de</strong>rada por número <strong>de</strong> genes validados <strong>de</strong> las AUCs provenientes <strong>de</strong><br />
las curvas ROC <strong>de</strong> los 15 pares <strong>de</strong> tejidos. Los dos métodos <strong>de</strong>sarrollados en este trabajo<br />
(ESLiMc y ESLiMt) muestran un mejor rendimiento que algoritmos FIRMA, ARH y COSIE.<br />
Las curvas ROC son una buena técnica para comparar métodos, ya que dan una visión global<br />
clasificando todos los datos disponibles (en este caso todos los genes). Sin embargo, al tratar<br />
con datos <strong>de</strong> genómicos, la dimensión <strong>de</strong> las matrices es muy alta y sólo una pequeña parte<br />
79