Alberto Risueño Pérez - Gredos - Universidad de Salamanca
Alberto Risueño Pérez - Gredos - Universidad de Salamanca
Alberto Risueño Pérez - Gredos - Universidad de Salamanca
You also want an ePaper? Increase the reach of your titles
YUMPU automatically turns print PDFs into web optimized ePapers that Google loves.
Capítulo 3<br />
muestran una señal clara que a<strong>de</strong>más es coherente con datos biológicos previamente<br />
publicados, en don<strong>de</strong> a RGN se le ha atribuido un evento <strong>de</strong> splicing diferencial entre los<br />
tejidos <strong>de</strong> hígado y cerebelo (Wang et al., 2008).<br />
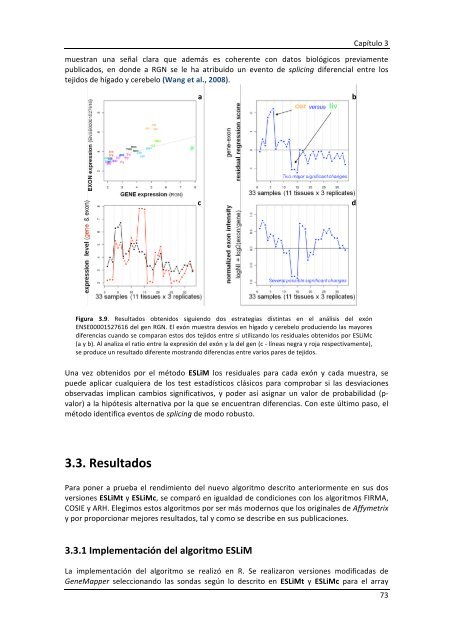
Figura 3.9. Resultados obtenidos siguiendo dos estrategias distintas en el análisis <strong>de</strong>l exón<br />
ENSE00001527616 <strong>de</strong>l gen RGN. El exón muestra <strong>de</strong>svíos en hígado y cerebelo produciendo las mayores<br />
diferencias cuando se comparan estos dos tejidos entre sí utilizando los residuales obtenidos por ESLiMc<br />
(a y b). Al analiza el ratio entre la expresión <strong>de</strong>l exón y la <strong>de</strong>l gen (c -‐ líneas negra y roja respectivamente),<br />
se produce un resultado diferente mostrando diferencias entre varios pares <strong>de</strong> tejidos.<br />
Una vez obtenidos por el método ESLiM los residuales para cada exón y cada muestra, se<br />
pue<strong>de</strong> aplicar cualquiera <strong>de</strong> los test estadísticos clásicos para comprobar si las <strong>de</strong>sviaciones<br />
observadas implican cambios significativos, y po<strong>de</strong>r así asignar un valor <strong>de</strong> probabilidad (p-‐<br />
valor) a la hipótesis alternativa por la que se encuentran diferencias. Con este último paso, el<br />
método i<strong>de</strong>ntifica eventos <strong>de</strong> splicing <strong>de</strong> modo robusto.<br />
3.3. Resultados<br />
Para poner a prueba el rendimiento <strong>de</strong>l nuevo algoritmo <strong>de</strong>scrito anteriormente en sus dos<br />
versiones ESLiMt y ESLiMc, se comparó en igualdad <strong>de</strong> condiciones con los algoritmos FIRMA,<br />
COSIE y ARH. Elegimos estos algoritmos por ser más mo<strong>de</strong>rnos que los originales <strong>de</strong> Affymetrix<br />
y por proporcionar mejores resultados, tal y como se <strong>de</strong>scribe en sus publicaciones.<br />
3.3.1 Implementación <strong>de</strong>l algoritmo ESLiM<br />
a b<br />
c d<br />
La implementación <strong>de</strong>l algoritmo se realizó en R. Se realizaron versiones modificadas <strong>de</strong><br />
GeneMapper seleccionando las sondas según lo <strong>de</strong>scrito en ESLiMt y ESLiMc para el array<br />
73