Alberto Risueño Pérez - Gredos - Universidad de Salamanca
Alberto Risueño Pérez - Gredos - Universidad de Salamanca
Alberto Risueño Pérez - Gredos - Universidad de Salamanca
You also want an ePaper? Increase the reach of your titles
YUMPU automatically turns print PDFs into web optimized ePapers that Google loves.
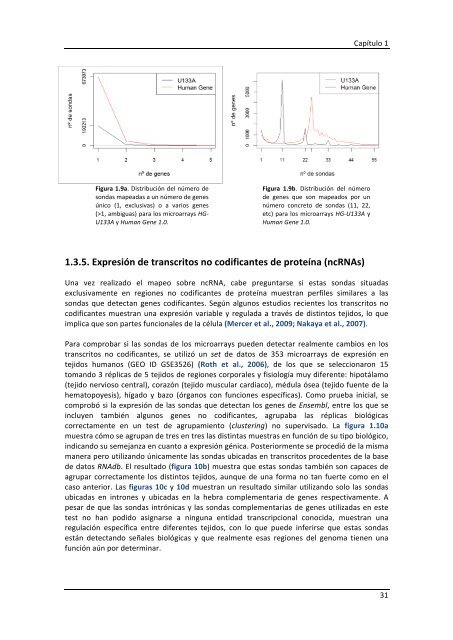
Figura 1.9a. Distribución <strong>de</strong>l número <strong>de</strong><br />
sondas mapeadas a un número <strong>de</strong> genes<br />
único (1, exclusivas) o a varios genes<br />
(>1, ambiguas) para los microarrays HG-‐<br />
U133A y Human Gene 1.0.<br />
1.3.5. Expresión <strong>de</strong> transcritos no codificantes <strong>de</strong> proteína (ncRNAs)<br />
Capítulo 1<br />
Figura 1.9b. Distribución <strong>de</strong>l número<br />
<strong>de</strong> genes que son mapeados por un<br />
número concreto <strong>de</strong> sondas (11, 22,<br />
etc) para los microarrays HG-‐U133A y<br />
Human Gene 1.0.<br />
Una vez realizado el mapeo sobre ncRNA, cabe preguntarse si estas sondas situadas<br />
exclusivamente en regiones no codificantes <strong>de</strong> proteína muestran perfiles similares a las<br />
sondas que <strong>de</strong>tectan genes codificantes. Según algunos estudios recientes los transcritos no<br />
codificantes muestran una expresión variable y regulada a través <strong>de</strong> distintos tejidos, lo que<br />
implica que son partes funcionales <strong>de</strong> la célula (Mercer et al., 2009; Nakaya et al., 2007).<br />
Para comprobar si las sondas <strong>de</strong> los microarrays pue<strong>de</strong>n <strong>de</strong>tectar realmente cambios en los<br />
transcritos no codificantes, se utilizó un set <strong>de</strong> datos <strong>de</strong> 353 microarrays <strong>de</strong> expresión en<br />
tejidos humanos (GEO ID GSE3526) (Roth et al., 2006), <strong>de</strong> los que se seleccionaron 15<br />
tomando 3 réplicas <strong>de</strong> 5 tejidos <strong>de</strong> regiones corporales y fisiología muy diferente: hipotálamo<br />
(tejido nervioso central), corazón (tejido muscular cardiaco), médula ósea (tejido fuente <strong>de</strong> la<br />
hematopoyesis), hígado y bazo (órganos con funciones específicas). Como prueba inicial, se<br />
comprobó si la expresión <strong>de</strong> las sondas que <strong>de</strong>tectan los genes <strong>de</strong> Ensembl, entre los que se<br />
incluyen también algunos genes no codificantes, agrupaba las réplicas biológicas<br />
correctamente en un test <strong>de</strong> agrupamiento (clustering) no supervisado. La figura 1.10a<br />
muestra cómo se agrupan <strong>de</strong> tres en tres las distintas muestras en función <strong>de</strong> su tipo biológico,<br />
indicando su semejanza en cuanto a expresión génica. Posteriormente se procedió <strong>de</strong> la misma<br />
manera pero utilizando únicamente las sondas ubicadas en transcritos proce<strong>de</strong>ntes <strong>de</strong> la base<br />
<strong>de</strong> datos RNAdb. El resultado (figura 10b) muestra que estas sondas también son capaces <strong>de</strong><br />
agrupar correctamente los distintos tejidos, aunque <strong>de</strong> una forma no tan fuerte como en el<br />
caso anterior. Las figuras 10c y 10d muestran un resultado similar utilizando solo las sondas<br />
ubicadas en intrones y ubicadas en la hebra complementaria <strong>de</strong> genes respectivamente. A<br />
pesar <strong>de</strong> que las sondas intrónicas y las sondas complementarias <strong>de</strong> genes utilizadas en este<br />
test no han podido asignarse a ninguna entidad transcripcional conocida, muestran una<br />
regulación específica entre diferentes tejidos, con lo que pue<strong>de</strong> inferirse que estas sondas<br />
están <strong>de</strong>tectando señales biológicas y que realmente esas regiones <strong>de</strong>l genoma tienen una<br />
función aún por <strong>de</strong>terminar.<br />
31