Alberto Risueño Pérez - Gredos - Universidad de Salamanca
Alberto Risueño Pérez - Gredos - Universidad de Salamanca
Alberto Risueño Pérez - Gredos - Universidad de Salamanca
You also want an ePaper? Increase the reach of your titles
YUMPU automatically turns print PDFs into web optimized ePapers that Google loves.
Capítulo 1<br />
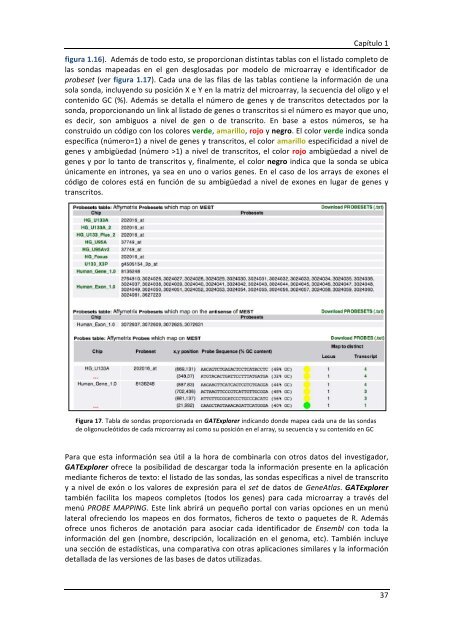
figura 1.16). A<strong>de</strong>más <strong>de</strong> todo esto, se proporcionan distintas tablas con el listado completo <strong>de</strong><br />
las sondas mapeadas en el gen <strong>de</strong>sglosadas por mo<strong>de</strong>lo <strong>de</strong> microarray e i<strong>de</strong>ntificador <strong>de</strong><br />
probeset (ver figura 1.17). Cada una <strong>de</strong> las filas <strong>de</strong> las tablas contiene la información <strong>de</strong> una<br />
sola sonda, incluyendo su posición X e Y en la matriz <strong>de</strong>l microarray, la secuencia <strong>de</strong>l oligo y el<br />
contenido GC (%). A<strong>de</strong>más se <strong>de</strong>talla el número <strong>de</strong> genes y <strong>de</strong> transcritos <strong>de</strong>tectados por la<br />
sonda, proporcionando un link al listado <strong>de</strong> genes o transcritos si el número es mayor que uno,<br />
es <strong>de</strong>cir, son ambiguos a nivel <strong>de</strong> gen o <strong>de</strong> transcrito. En base a estos números, se ha<br />
construido un código con los colores ver<strong>de</strong>, amarillo, rojo y negro. El color ver<strong>de</strong> indica sonda<br />
específica (número=1) a nivel <strong>de</strong> genes y transcritos, el color amarillo especificidad a nivel <strong>de</strong><br />
genes y ambigüedad (número >1) a nivel <strong>de</strong> transcritos, el color rojo ambigüedad a nivel <strong>de</strong><br />
genes y por lo tanto <strong>de</strong> transcritos y, finalmente, el color negro indica que la sonda se ubica<br />
únicamente en intrones, ya sea en uno o varios genes. En el caso <strong>de</strong> los arrays <strong>de</strong> exones el<br />
código <strong>de</strong> colores está en función <strong>de</strong> su ambigüedad a nivel <strong>de</strong> exones en lugar <strong>de</strong> genes y<br />
transcritos.<br />
Figura 17. Tabla <strong>de</strong> sondas proporcionada en GATExplorer indicando don<strong>de</strong> mapea cada una <strong>de</strong> las sondas<br />
<strong>de</strong> oligonucleótidos <strong>de</strong> cada microarray así como su posición en el array, su secuencia y su contenido en GC<br />
Para que esta información sea útil a la hora <strong>de</strong> combinarla con otros datos <strong>de</strong>l investigador,<br />
GATExplorer ofrece la posibilidad <strong>de</strong> <strong>de</strong>scargar toda la información presente en la aplicación<br />
mediante ficheros <strong>de</strong> texto: el listado <strong>de</strong> las sondas, las sondas específicas a nivel <strong>de</strong> transcrito<br />
y a nivel <strong>de</strong> exón o los valores <strong>de</strong> expresión para el set <strong>de</strong> datos <strong>de</strong> GeneAtlas. GATExplorer<br />
también facilita los mapeos completos (todos los genes) para cada microarray a través <strong>de</strong>l<br />
menú PROBE MAPPING. Este link abrirá un pequeño portal con varias opciones en un menú<br />
lateral ofreciendo los mapeos en dos formatos, ficheros <strong>de</strong> texto o paquetes <strong>de</strong> R. A<strong>de</strong>más<br />
ofrece unos ficheros <strong>de</strong> anotación para asociar cada i<strong>de</strong>ntificador <strong>de</strong> Ensembl con toda la<br />
información <strong>de</strong>l gen (nombre, <strong>de</strong>scripción, localización en el genoma, etc). También incluye<br />
una sección <strong>de</strong> estadísticas, una comparativa con otras aplicaciones similares y la información<br />
<strong>de</strong>tallada <strong>de</strong> las versiones <strong>de</strong> las bases <strong>de</strong> datos utilizadas.<br />
37