Alberto Risueño Pérez - Gredos - Universidad de Salamanca
Alberto Risueño Pérez - Gredos - Universidad de Salamanca
Alberto Risueño Pérez - Gredos - Universidad de Salamanca
You also want an ePaper? Increase the reach of your titles
YUMPU automatically turns print PDFs into web optimized ePapers that Google loves.
Capítulo 2<br />
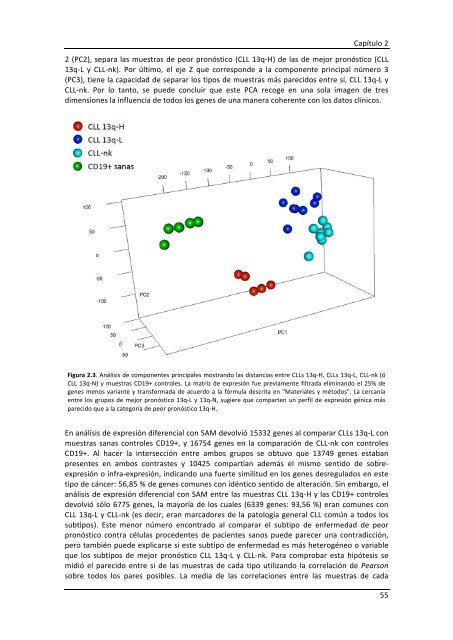
2 (PC2), separa las muestras <strong>de</strong> peor pronóstico (CLL 13q-‐H) <strong>de</strong> las <strong>de</strong> mejor pronóstico (CLL<br />
13q-‐L y CLL-‐nk). Por último, el eje Z que correspon<strong>de</strong> a la componente principal número 3<br />
(PC3), tiene la capacidad <strong>de</strong> separar los tipos <strong>de</strong> muestras más parecidos entre sí, CLL 13q-‐L y<br />
CLL-‐nk. Por lo tanto, se pue<strong>de</strong> concluir que este PCA recoge en una sola imagen <strong>de</strong> tres<br />
dimensiones la influencia <strong>de</strong> todos los genes <strong>de</strong> una manera coherente con los datos clínicos.<br />
Figura 2.3. Análisis <strong>de</strong> componentes principales mostrando las distancias entre CLLs 13q-‐H, CLLs 13q-‐L, CLL-‐nk (ó<br />
CLL 13q-‐N) y muestras CD19+ controles. La matriz <strong>de</strong> expresión fue previamente filtrada eliminando el 25% <strong>de</strong><br />
genes menos variante y transformada <strong>de</strong> acuerdo a la fórmula <strong>de</strong>scrita en "Materiales y métodos". La cercanía<br />
entre los grupos <strong>de</strong> mejor pronóstico 13q-‐L y 13q-‐N, sugiere que comparten un perfil <strong>de</strong> expresión génica más<br />
parecido que a la categoría <strong>de</strong> peor pronóstico 13q-‐H.<br />
En análisis <strong>de</strong> expresión diferencial con SAM <strong>de</strong>volvió 15332 genes al comparar CLLs 13q-‐L con<br />
muestras sanas controles CD19+, y 16754 genes en la comparación <strong>de</strong> CLL-‐nk con controles<br />
CD19+. Al hacer la intersección entre ambos grupos se obtuvo que 13749 genes estaban<br />
presentes en ambos contrastes y 10425 compartían a<strong>de</strong>más el mismo sentido <strong>de</strong> sobre-‐<br />
expresión o infra-‐expresión, indicando una fuerte similitud en los genes <strong>de</strong>sregulados en este<br />
tipo <strong>de</strong> cáncer: 56,85 % <strong>de</strong> genes comunes con idéntico sentido <strong>de</strong> alteración. Sin embargo, el<br />
análisis <strong>de</strong> expresión diferencial con SAM entre las muestras CLL 13q-‐H y las CD19+ controles<br />
<strong>de</strong>volvió sólo 6775 genes, la mayoría <strong>de</strong> los cuales (6339 genes: 93,56 %) eran comunes con<br />
CLL 13q-‐L y CLL-‐nk (es <strong>de</strong>cir, eran marcadores <strong>de</strong> la patología general CLL común a todos los<br />
subtipos). Este menor número encontrado al comparar el subtipo <strong>de</strong> enfermedad <strong>de</strong> peor<br />
pronóstico contra células proce<strong>de</strong>ntes <strong>de</strong> pacientes sanos pue<strong>de</strong> parecer una contradicción,<br />
pero también pue<strong>de</strong> explicarse si este subtipo <strong>de</strong> enfermedad es más heterogéneo o variable<br />
que los subtipos <strong>de</strong> mejor pronóstico CLL 13q-‐L y CLL-‐nk. Para comprobar esta hipótesis se<br />
midió el parecido entre sí <strong>de</strong> las muestras <strong>de</strong> cada tipo utilizando la correlación <strong>de</strong> Pearson<br />
sobre todos los pares posibles. La media <strong>de</strong> las correlaciones entre las muestras <strong>de</strong> cada<br />
55