Alberto Risueño Pérez - Gredos - Universidad de Salamanca
Alberto Risueño Pérez - Gredos - Universidad de Salamanca
Alberto Risueño Pérez - Gredos - Universidad de Salamanca
Create successful ePaper yourself
Turn your PDF publications into a flip-book with our unique Google optimized e-Paper software.
Capítulo 3<br />
métodos analizados produciendo un total <strong>de</strong> 15 contrastes. Los algoritmos ESLiMc, ESLiMt,<br />
FIRMA y COSIE <strong>de</strong>vuelven un valor por muestra y exón, por lo que solamente hizo falta<br />
ejecutar una vez cada uno <strong>de</strong> ellos utilizando posteriormente el paquete limma (Smyth et al.,<br />
2012) para realizar el ranking <strong>de</strong> exones por cada par <strong>de</strong> tejidos. ARH <strong>de</strong>vuelve directamente<br />
un valor <strong>de</strong> significación por gen (ARH values), por lo que <strong>de</strong>bió ejecutarse 15 veces, uno por<br />
cada contraste.<br />
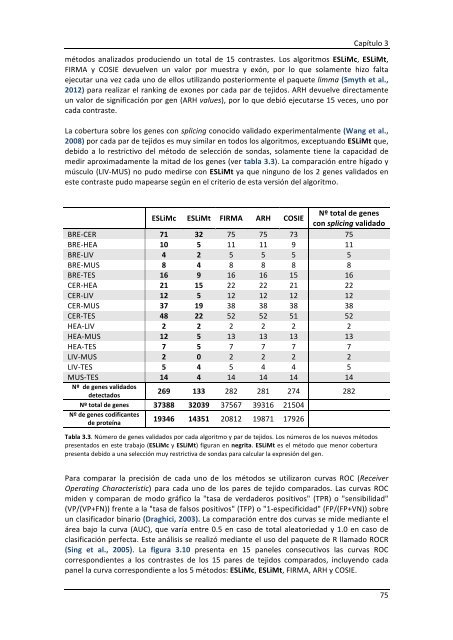
La cobertura sobre los genes con splicing conocido validado experimentalmente (Wang et al.,<br />
2008) por cada par <strong>de</strong> tejidos es muy similar en todos los algoritmos, exceptuando ESLiMt que,<br />
<strong>de</strong>bido a lo restrictivo <strong>de</strong>l método <strong>de</strong> selección <strong>de</strong> sondas, solamente tiene la capacidad <strong>de</strong><br />
medir aproximadamente la mitad <strong>de</strong> los genes (ver tabla 3.3). La comparación entre hígado y<br />
músculo (LIV-‐MUS) no pudo medirse con ESLiMt ya que ninguno <strong>de</strong> los 2 genes validados en<br />
este contraste pudo mapearse según en el criterio <strong>de</strong> esta versión <strong>de</strong>l algoritmo.<br />
ESLiMc ESLiMt FIRMA ARH COSIE<br />
Nº total <strong>de</strong> genes<br />
con splicing validado<br />
BRE-‐CER 71 32 75 75 73 75<br />
BRE-‐HEA 10 5 11 11 9 11<br />
BRE-‐LIV 4 2 5 5 5 5<br />
BRE-‐MUS 8 4 8 8 8 8<br />
BRE-‐TES 16 9 16 16 15 16<br />
CER-‐HEA 21 15 22 22 21 22<br />
CER-‐LIV 12 5 12 12 12 12<br />
CER-‐MUS 37 19 38 38 38 38<br />
CER-‐TES 48 22 52 52 51 52<br />
HEA-‐LIV 2 2 2 2 2 2<br />
HEA-‐MUS 12 5 13 13 13 13<br />
HEA-‐TES 7 5 7 7 7 7<br />
LIV-‐MUS 2 0 2 2 2 2<br />
LIV-‐TES 5 4 5 4 4 5<br />
MUS-‐TES 14 4 14 14 14 14<br />
Nº <strong>de</strong> genes validados<br />
<strong>de</strong>tectados<br />
269 133 282 281 274 282<br />
Nº total <strong>de</strong> genes 37388 32039 37567 39316 21504<br />
Nº <strong>de</strong> genes codificantes<br />
<strong>de</strong> proteína<br />
19346 14351 20812 19871 17926<br />
Tabla 3.3. Número <strong>de</strong> genes validados por cada algoritmo y par <strong>de</strong> tejidos. Los números <strong>de</strong> los nuevos métodos<br />
presentados en este trabajo (ESLiMc y ESLiMt) figuran en negrita. ESLiMt es el método que menor cobertura<br />
presenta <strong>de</strong>bido a una selección muy restrictiva <strong>de</strong> sondas para calcular la expresión <strong>de</strong>l gen.<br />
Para comparar la precisión <strong>de</strong> cada uno <strong>de</strong> los métodos se utilizaron curvas ROC (Receiver<br />
Operating Characteristic) para cada uno <strong>de</strong> los pares <strong>de</strong> tejido comparados. Las curvas ROC<br />
mi<strong>de</strong>n y comparan <strong>de</strong> modo gráfico la "tasa <strong>de</strong> verda<strong>de</strong>ros positivos" (TPR) o "sensibilidad"<br />
(VP/(VP+FN)) frente a la "tasa <strong>de</strong> falsos positivos" (TFP) o "1-‐especificidad" (FP/(FP+VN)) sobre<br />
un clasificador binario (Draghici, 2003). La comparación entre dos curvas se mi<strong>de</strong> mediante el<br />
área bajo la curva (AUC), que varía entre 0.5 en caso <strong>de</strong> total aleatoriedad y 1.0 en caso <strong>de</strong><br />
clasificación perfecta. Este análisis se realizó mediante el uso <strong>de</strong>l paquete <strong>de</strong> R llamado ROCR<br />
(Sing et al., 2005). La figura 3.10 presenta en 15 paneles consecutivos las curvas ROC<br />
correspondientes a los contrastes <strong>de</strong> los 15 pares <strong>de</strong> tejidos comparados, incluyendo cada<br />
panel la curva correspondiente a los 5 métodos: ESLiMc, ESLiMt, FIRMA, ARH y COSIE.<br />
75