Alberto Risueño Pérez - Gredos - Universidad de Salamanca
Alberto Risueño Pérez - Gredos - Universidad de Salamanca
Alberto Risueño Pérez - Gredos - Universidad de Salamanca
You also want an ePaper? Increase the reach of your titles
YUMPU automatically turns print PDFs into web optimized ePapers that Google loves.
Tesis Doctoral<br />
3.8.b). Sin embargo la expresión global <strong>de</strong>l gen MEST sufre gran<strong>de</strong>s variaciones en los distintos<br />
tejidos (figura 3.8.c), afectando a la relación <strong>de</strong> expresión entre gen y exón (figura 3.8.d). Ante<br />
esto, cualquier estrategia <strong>de</strong> análisis <strong>de</strong> splicing que no tenga en cuenta el <strong>de</strong>sacuerdo en la<br />
dinámica <strong>de</strong> expresión <strong>de</strong> las distintas entida<strong>de</strong>s transcripcionales (en este caso entre el exón y<br />
el gen), estará expuesta a la <strong>de</strong>tección <strong>de</strong> falsos positivos.<br />
72<br />
a b<br />
c d<br />
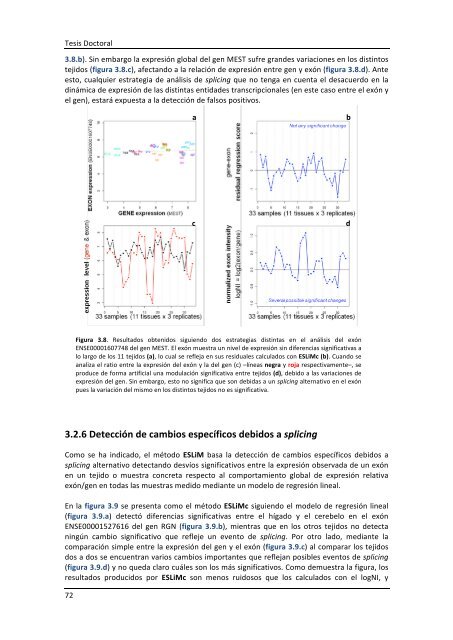
Figura 3.8. Resultados obtenidos siguiendo dos estrategias distintas en el análisis <strong>de</strong>l exón<br />
ENSE00001607748 <strong>de</strong>l gen MEST. El exón muestra un nivel <strong>de</strong> expresión sin diferencias significativas a<br />
lo largo <strong>de</strong> los 11 tejidos (a), lo cual se refleja en sus residuales calculados con ESLiMc (b). Cuando se<br />
analiza el ratio entre la expresión <strong>de</strong>l exón y la <strong>de</strong>l gen (c) –líneas negra y roja respectivamente–, se<br />
produce <strong>de</strong> forma artificial una modulación significativa entre tejidos (d), <strong>de</strong>bido a las variaciones <strong>de</strong><br />
expresión <strong>de</strong>l gen. Sin embargo, esto no significa que son <strong>de</strong>bidas a un splicing alternativo en el exón<br />
pues la variación <strong>de</strong>l mismo en los distintos tejidos no es significativa.<br />
3.2.6 Detección <strong>de</strong> cambios específicos <strong>de</strong>bidos a splicing<br />
Como se ha indicado, el método ESLiM basa la <strong>de</strong>tección <strong>de</strong> cambios específicos <strong>de</strong>bidos a<br />
splicing alternativo <strong>de</strong>tectando <strong>de</strong>svíos significativos entre la expresión observada <strong>de</strong> un exón<br />
en un tejido o muestra concreta respecto al comportamiento global <strong>de</strong> expresión relativa<br />
exón/gen en todas las muestras medido mediante un mo<strong>de</strong>lo <strong>de</strong> regresión lineal.<br />
En la figura 3.9 se presenta como el método ESLiMc siguiendo el mo<strong>de</strong>lo <strong>de</strong> regresión lineal<br />
(figura 3.9.a) <strong>de</strong>tectó diferencias significativas entre el hígado y el cerebelo en el exón<br />
ENSE00001527616 <strong>de</strong>l gen RGN (figura 3.9.b), mientras que en los otros tejidos no <strong>de</strong>tecta<br />
ningún cambio significativo que refleje un evento <strong>de</strong> splicing. Por otro lado, mediante la<br />
comparación simple entre la expresión <strong>de</strong>l gen y el exón (figura 3.9.c) al comparar los tejidos<br />
dos a dos se encuentran varios cambios importantes que reflejan posibles eventos <strong>de</strong> splicing<br />
(figura 3.9.d) y no queda claro cuáles son los más significativos. Como <strong>de</strong>muestra la figura, los<br />
resultados producidos por ESLiMc son menos ruidosos que los calculados con el logNI, y