Alberto Risueño Pérez - Gredos - Universidad de Salamanca
Alberto Risueño Pérez - Gredos - Universidad de Salamanca
Alberto Risueño Pérez - Gredos - Universidad de Salamanca
You also want an ePaper? Increase the reach of your titles
YUMPU automatically turns print PDFs into web optimized ePapers that Google loves.
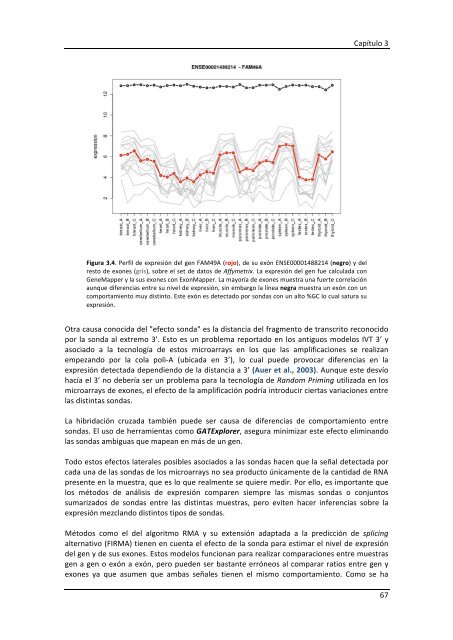
Figura 3.4. Perfil <strong>de</strong> expresión <strong>de</strong>l gen FAM49A (rojo), <strong>de</strong> su exón ENSE00001488214 (negro) y <strong>de</strong>l<br />
resto <strong>de</strong> exones (gris), sobre el set <strong>de</strong> datos <strong>de</strong> Affymetrix. La expresión <strong>de</strong>l gen fue calculada con<br />
GeneMapper y la sus exones con ExonMapper. La mayoría <strong>de</strong> exones muestra una fuerte correlación<br />
aunque diferencias entre su nivel <strong>de</strong> expresión, sin embargo la línea negra muestra un exón con un<br />
comportamiento muy distinto. Este exón es <strong>de</strong>tectado por sondas con un alto %GC lo cual satura su<br />
expresión.<br />
Capítulo 3<br />
Otra causa conocida <strong>de</strong>l "efecto sonda" es la distancia <strong>de</strong>l fragmento <strong>de</strong> transcrito reconocido<br />
por la sonda al extremo 3’. Esto es un problema reportado en los antiguos mo<strong>de</strong>los IVT 3’ y<br />
asociado a la tecnología <strong>de</strong> estos microarrays en los que las amplificaciones se realizan<br />
empezando por la cola poli-‐A (ubicada en 3’), lo cual pue<strong>de</strong> provocar diferencias en la<br />
expresión <strong>de</strong>tectada <strong>de</strong>pendiendo <strong>de</strong> la distancia a 3’ (Auer et al., 2003). Aunque este <strong>de</strong>svío<br />
hacía el 3’ no <strong>de</strong>bería ser un problema para la tecnología <strong>de</strong> Random Priming utilizada en los<br />
microarrays <strong>de</strong> exones, el efecto <strong>de</strong> la amplificación podría introducir ciertas variaciones entre<br />
las distintas sondas.<br />
La hibridación cruzada también pue<strong>de</strong> ser causa <strong>de</strong> diferencias <strong>de</strong> comportamiento entre<br />
sondas. El uso <strong>de</strong> herramientas como GATExplorer, asegura minimizar este efecto eliminando<br />
las sondas ambiguas que mapean en más <strong>de</strong> un gen.<br />
Todo estos efectos laterales posibles asociados a las sondas hacen que la señal <strong>de</strong>tectada por<br />
cada una <strong>de</strong> las sondas <strong>de</strong> los microarrays no sea producto únicamente <strong>de</strong> la cantidad <strong>de</strong> RNA<br />
presente en la muestra, que es lo que realmente se quiere medir. Por ello, es importante que<br />
los métodos <strong>de</strong> análisis <strong>de</strong> expresión comparen siempre las mismas sondas o conjuntos<br />
sumarizados <strong>de</strong> sondas entre las distintas muestras, pero eviten hacer inferencias sobre la<br />
expresión mezclando distintos tipos <strong>de</strong> sondas.<br />
Métodos como el <strong>de</strong>l algoritmo RMA y su extensión adaptada a la predicción <strong>de</strong> splicing<br />
alternativo (FIRMA) tienen en cuenta el efecto <strong>de</strong> la sonda para estimar el nivel <strong>de</strong> expresión<br />
<strong>de</strong>l gen y <strong>de</strong> sus exones. Estos mo<strong>de</strong>los funcionan para realizar comparaciones entre muestras<br />
gen a gen o exón a exón, pero pue<strong>de</strong>n ser bastante erróneos al comparar ratios entre gen y<br />
exones ya que asumen que ambas señales tienen el mismo comportamiento. Como se ha<br />
67