Alberto Risueño Pérez - Gredos - Universidad de Salamanca
Alberto Risueño Pérez - Gredos - Universidad de Salamanca
Alberto Risueño Pérez - Gredos - Universidad de Salamanca
You also want an ePaper? Increase the reach of your titles
YUMPU automatically turns print PDFs into web optimized ePapers that Google loves.
Tesis Doctoral<br />
<strong>de</strong>l inicio <strong>de</strong>l ranking por significación será consi<strong>de</strong>rada por el investigador. Esto significa que la<br />
información realmente útil es la proporcionada a tasas bajas <strong>de</strong> falsos positivos. Por este<br />
motivo hemos hecho un estudio <strong>de</strong> la cantidad <strong>de</strong> verda<strong>de</strong>ros positivos eligiendo un criterio<br />
fijo para todos los métodos y pares <strong>de</strong> tejidos, como es consi<strong>de</strong>rar únicamente los primeros n<br />
genes más significativos eligiendo un umbral arbitrario <strong>de</strong> 1000 genes. Los resultados fueron<br />
diferentes a los obtenidos con las curvas ROC. En este caso los métodos ESLiMc y ESLiMt<br />
mostraron un comportamiento bastante distinto entre si, encontrando 61 y 29 genes<br />
respectivamente. El or<strong>de</strong>n entre FIRMA, ARH y COSIE también se alteró, mostrando COSIE<br />
mostró mejor resultado que los otros dos métodos (ver tabla 3.5 y figura 3.12).<br />
80<br />
ESLiMc ESLiMt FIRMA ARH COSIE<br />
BRE-‐CER 17 8 6 6 10<br />
BRE-‐HEA 2 0 2 1 2<br />
BRE-‐LIV 1 0 0 2 1<br />
BRE-‐MUS 3 1 2 3 2<br />
BRE-‐TES 1 1 0 0 0<br />
CER-‐HEA 1 3 1 1 2<br />
CER-‐LIV 1 1 1 0 1<br />
CER-‐MUS 10 3 3 5 6<br />
CER-‐TES 10 5 1 4 3<br />
HEA-‐LIV 0 0 0 0 0<br />
HEA-‐MUS 5 2 1 0 3<br />
HEA-‐TES 4 3 3 1 4<br />
LIV-‐MUS 1 0 0 0 1<br />
LIV-‐TES 0 0 1 1 0<br />
MUS-‐TES 5 2 3 6 5<br />
Suma 61 29 24 30 40<br />
Tabla 3.5. Número <strong>de</strong> genes con splicing validados en las distintas comparaciones entre tejido <strong>de</strong>tectados en los<br />
1000 genes más significativos. Se comparan 5 métodos <strong>de</strong> <strong>de</strong>tección <strong>de</strong> splicing en 15 comparaciones binarias.<br />
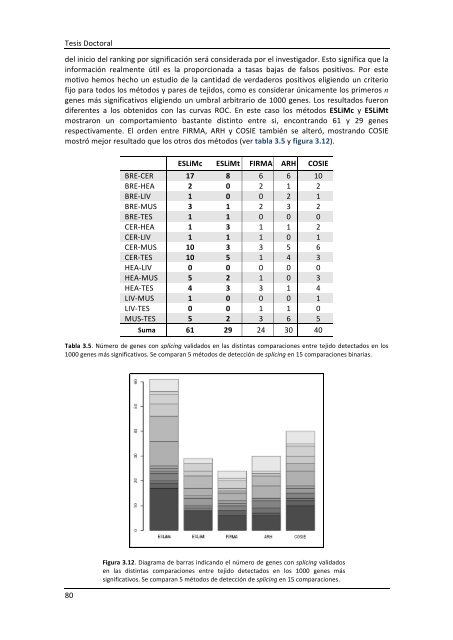
Figura 3.12. Diagrama <strong>de</strong> barras indicando el número <strong>de</strong> genes con splicing validados<br />
en las distintas comparaciones entre tejido <strong>de</strong>tectados en los 1000 genes más<br />
significativos. Se comparan 5 métodos <strong>de</strong> <strong>de</strong>tección <strong>de</strong> splicing en 15 comparaciones.