Alberto Risueño Pérez - Gredos - Universidad de Salamanca
Alberto Risueño Pérez - Gredos - Universidad de Salamanca
Alberto Risueño Pérez - Gredos - Universidad de Salamanca
Create successful ePaper yourself
Turn your PDF publications into a flip-book with our unique Google optimized e-Paper software.
Tesis Doctoral<br />
dicho, la comparación <strong>de</strong> entida<strong>de</strong>s <strong>de</strong>tectadas por distintas sondas pue<strong>de</strong> ser bastante<br />
problemática y el único algoritmo reportado diseñado para afrontar esta dificultad es COSIE<br />
(Gaidatzis et al., 2009).<br />
3.2.3 Un nuevo método <strong>de</strong> análisis <strong>de</strong> splicing: Exon Splicing using Linear<br />
Mo<strong>de</strong>ling (ESLiM)<br />
Como se ha <strong>de</strong>scrito, el "efecto sonda" pue<strong>de</strong> tener un peso gran<strong>de</strong> en los datos proce<strong>de</strong>ntes<br />
<strong>de</strong> microarrays, por ello, las alteraciones que pueda producir <strong>de</strong>berían <strong>de</strong> ser tenidas en<br />
cuenta a la hora <strong>de</strong> analizar e interpretar datos <strong>de</strong> expresión. Sin embargo, esto no parece<br />
haber sido <strong>de</strong>bidamente tratado en las publicaciones previas en el ámbito <strong>de</strong> <strong>de</strong>tección <strong>de</strong><br />
splicing alternativo con arrays <strong>de</strong> exones. El diseño <strong>de</strong> una estrategia para corregir dicho<br />
problema podría ayudar a mejorar los resultados obtenidos con este tipo <strong>de</strong> tecnología y ha<br />
sido la principal motivación para la realización <strong>de</strong> esta parte <strong>de</strong>l presente trabajo <strong>de</strong> Tesis<br />
Doctoral.<br />
La figura 3.1 puso <strong>de</strong> manifiesto cómo las sondas reaccionan <strong>de</strong> distinta forma ante<br />
concentraciones crecientes <strong>de</strong> RNA mostrando distintas pendientes. De la misma forma, se<br />
pue<strong>de</strong> tratar <strong>de</strong> hacer una estimación <strong>de</strong>l efecto exón para la <strong>de</strong>tección <strong>de</strong> splicing alternativo.<br />
Dado que en un set <strong>de</strong> datos cualquiera a analizar, no se pue<strong>de</strong> conocer la cantidad exacta <strong>de</strong><br />
RNA <strong>de</strong> cada exón presente en cada una <strong>de</strong> las muestras, este dato se pue<strong>de</strong> aproximar<br />
mediante la expresión global <strong>de</strong>l gen.<br />
68<br />
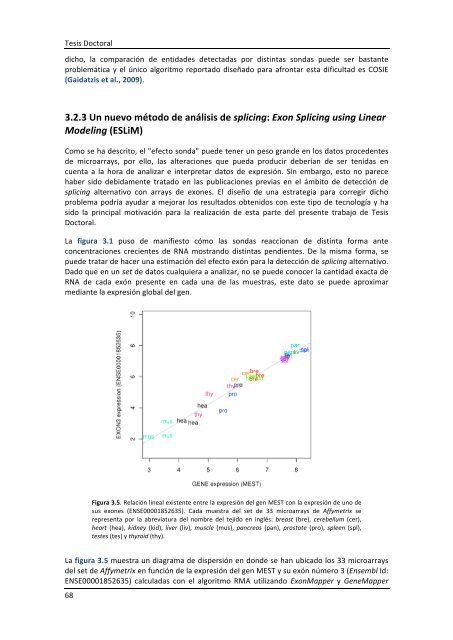
Figura 3.5. Relación lineal existente entre la expresión <strong>de</strong>l gen MEST con la expresión <strong>de</strong> uno <strong>de</strong><br />
sus exones (ENSE00001852635). Cada muestra <strong>de</strong>l set <strong>de</strong> 33 microarrays <strong>de</strong> Affymetrix se<br />
representa por la abreviatura <strong>de</strong>l nombre <strong>de</strong>l tejido en inglés: breast (bre), cerebellum (cer),<br />
heart (hea), kidney (kid), liver (liv), muscle (mus), pancreas (pan), prostate (pro), spleen (spl),<br />
testes (tes) y thyroid (thy).<br />
La figura 3.5 muestra un diagrama <strong>de</strong> dispersión en don<strong>de</strong> se han ubicado los 33 microarrays<br />
<strong>de</strong>l set <strong>de</strong> Affymetrix en función <strong>de</strong> la expresión <strong>de</strong>l gen MEST y su exón número 3 (Ensembl Id:<br />
ENSE00001852635) calculadas con el algoritmo RMA utilizando ExonMapper y GeneMapper