Mechanistische Analysen zu Krankheits-korrelierten SNPs in ...
Mechanistische Analysen zu Krankheits-korrelierten SNPs in ...
Mechanistische Analysen zu Krankheits-korrelierten SNPs in ...
Erfolgreiche ePaper selbst erstellen
Machen Sie aus Ihren PDF Publikationen ein blätterbares Flipbook mit unserer einzigartigen Google optimierten e-Paper Software.
5.6 Untersuchungen im MRAS-System<br />
110<br />
100<br />
94<br />
*<br />
86<br />
90<br />
***<br />
90<br />
FL/RL <strong>in</strong> %<br />
80<br />
70<br />
75<br />
MRAS 3'-UTR C<br />
MRAS 3'-UTR U<br />
60<br />
50<br />
miR-195<br />
miR-135a<br />
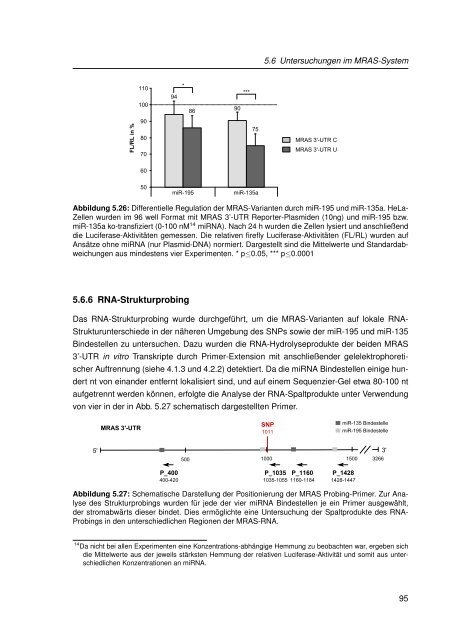
Abbildung 5.26: Differentielle Regulation der MRAS-Varianten durch miR-195 und miR-135a. HeLa-<br />
Zellen wurden im 96 well Format mit MRAS 3’-UTR Reporter-Plasmiden (10ng) und miR-195 bzw.<br />
miR-135a ko-transfiziert (0-100 nM 14 miRNA). Nach 24 h wurden die Zellen lysiert und anschließend<br />
die Luciferase-Aktivitäten gemessen. Die relativen firefly Luciferase-Aktivitäten (FL/RL) wurden auf<br />
Ansätze ohne miRNA (nur Plasmid-DNA) normiert. Dargestellt s<strong>in</strong>d die Mittelwerte und Standardabweichungen<br />
aus m<strong>in</strong>destens vier Experimenten. * p≤0.05, *** p≤0.0001<br />
5.6.6 RNA-Strukturprob<strong>in</strong>g<br />
Das RNA-Strukturprob<strong>in</strong>g wurde durchgeführt, um die MRAS-Varianten auf lokale RNA-<br />
Strukturunterschiede <strong>in</strong> der näheren Umgebung des <strong>SNPs</strong> sowie der miR-195 und miR-135<br />
B<strong>in</strong>destellen <strong>zu</strong> untersuchen. Da<strong>zu</strong> wurden die RNA-Hydrolyseprodukte der beiden MRAS<br />
3’-UTR <strong>in</strong> vitro Transkripte durch Primer-Extension mit anschließender gelelektrophoretischer<br />
Auftrennung (siehe 4.1.3 und 4.2.2) detektiert. Da die miRNA B<strong>in</strong>destellen e<strong>in</strong>ige hundert<br />
nt von e<strong>in</strong>ander entfernt lokalisiert s<strong>in</strong>d, und auf e<strong>in</strong>em Sequenzier-Gel etwa 80-100 nt<br />
aufgetrennt werden können, erfolgte die Analyse der RNA-Spaltprodukte unter Verwendung<br />
von vier <strong>in</strong> der <strong>in</strong> Abb. 5.27 schematisch dargestellten Primer.<br />
MRAS 3′-UTR<br />
SNP<br />
1011<br />
miR-135 B<strong>in</strong>destelle<br />
miR-195 B<strong>in</strong>destelle<br />
5'<br />
500 1000 1500<br />
3266<br />
3'<br />
P_400<br />
400-420<br />
P_1035 P_1160<br />
1035-1055 1160-1184<br />
P_1428<br />
1428-1447<br />
Abbildung 5.27: Schematische Darstellung der Positionierung der MRAS Prob<strong>in</strong>g-Primer. Zur Analyse<br />
des Strukturprob<strong>in</strong>gs wurden für jede der vier miRNA B<strong>in</strong>destellen je e<strong>in</strong> Primer ausgewählt,<br />
der stromabwärts dieser b<strong>in</strong>det. Dies ermöglichte e<strong>in</strong>e Untersuchung der Spaltprodukte des RNA-<br />
Prob<strong>in</strong>gs <strong>in</strong> den unterschiedlichen Regionen der MRAS-RNA.<br />
14 Da nicht bei allen Experimenten e<strong>in</strong>e Konzentrations-abhängige Hemmung <strong>zu</strong> beobachten war, ergeben sich<br />
die Mittelwerte aus der jeweils stärksten Hemmung der relativen Luciferase-Aktivität und somit aus unterschiedlichen<br />
Konzentrationen an miRNA.<br />
95