Mechanistische Analysen zu Krankheits-korrelierten SNPs in ...
Mechanistische Analysen zu Krankheits-korrelierten SNPs in ...
Mechanistische Analysen zu Krankheits-korrelierten SNPs in ...
Erfolgreiche ePaper selbst erstellen
Machen Sie aus Ihren PDF Publikationen ein blätterbares Flipbook mit unserer einzigartigen Google optimierten e-Paper Software.
5 Ergebnisse<br />
A<br />
Pb 2+<br />
B<br />
C<br />
RNase T1<br />
TCF21 3′-UTR<br />
C<br />
- -<br />
G<br />
A C G T<br />
TCF21 3′-UTR<br />
C<br />
- -<br />
G<br />
A 1045<br />
G 1046<br />
A 1047<br />
A 1048<br />
C 1049<br />
U 1059<br />
U 1060<br />
C 1061<br />
A 1062<br />
U 1063<br />
C 1068<br />
U 1069<br />
C 1072<br />
U 1073<br />
C/G 1058<br />
G 1041<br />
G 1046<br />
G 1053<br />
G 1054<br />
G 1056<br />
C/G 1058<br />
G 1070<br />
D<br />
rs12190287<br />
TCF21 C<br />
TCF21 G<br />
GGCUGAGAACUUCGGUGACUUCAUCCACCUGUCUAU<br />
1040<br />
1050<br />
1060 1070<br />
GGCUGAGAACUUCGGUGAGUUCAUCCACCUGUCUAU<br />
Pb 2+<br />
stark<br />
mittel<br />
schwach<br />
T1<br />
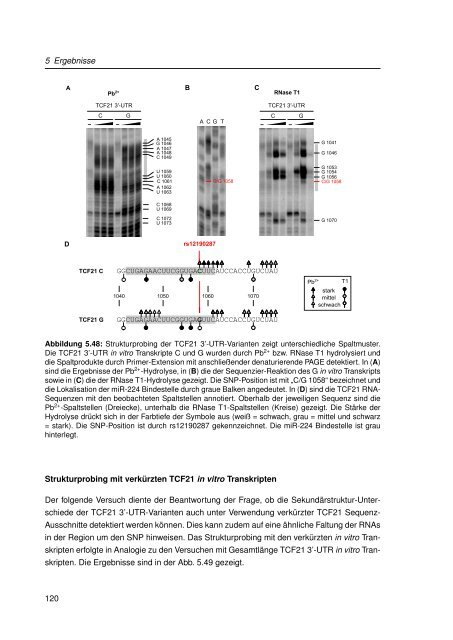
Abbildung 5.48: Strukturprob<strong>in</strong>g der TCF21 3’-UTR-Varianten zeigt unterschiedliche Spaltmuster.<br />
Die TCF21 3’-UTR <strong>in</strong> vitro Transkripte C und G wurden durch Pb 2+ bzw. RNase T1 hydrolysiert und<br />
die Spaltprodukte durch Primer-Extension mit anschließender denaturierende PAGE detektiert. In (A)<br />
s<strong>in</strong>d die Ergebnisse der Pb 2+ -Hydrolyse, <strong>in</strong> (B) die der Sequenzier-Reaktion des G <strong>in</strong> vitro Transkripts<br />
sowie <strong>in</strong> (C) die der RNase T1-Hydrolyse gezeigt. Die SNP-Position ist mit „C/G 1058“ bezeichnet und<br />
die Lokalisation der miR-224 B<strong>in</strong>destelle durch graue Balken angedeutet. In (D) s<strong>in</strong>d die TCF21 RNA-<br />
Sequenzen mit den beobachteten Spaltstellen annotiert. Oberhalb der jeweiligen Sequenz s<strong>in</strong>d die<br />
Pb 2+ -Spaltstellen (Dreiecke), unterhalb die RNase T1-Spaltstellen (Kreise) gezeigt. Die Stärke der<br />
Hydrolyse drückt sich <strong>in</strong> der Farbtiefe der Symbole aus (weiß = schwach, grau = mittel und schwarz<br />
= stark). Die SNP-Position ist durch rs12190287 gekennzeichnet. Die miR-224 B<strong>in</strong>destelle ist grau<br />
h<strong>in</strong>terlegt.<br />
Strukturprob<strong>in</strong>g mit verkürzten TCF21 <strong>in</strong> vitro Transkripten<br />
Der folgende Versuch diente der Beantwortung der Frage, ob die Sekundärstruktur-Unterschiede<br />
der TCF21 3’-UTR-Varianten auch unter Verwendung verkürzter TCF21 Sequenz-<br />
Ausschnitte detektiert werden können. Dies kann <strong>zu</strong>dem auf e<strong>in</strong>e ähnliche Faltung der RNAs<br />
<strong>in</strong> der Region um den SNP h<strong>in</strong>weisen. Das Strukturprob<strong>in</strong>g mit den verkürzten <strong>in</strong> vitro Transkripten<br />
erfolgte <strong>in</strong> Analogie <strong>zu</strong> den Versuchen mit Gesamtlänge TCF21 3’-UTR <strong>in</strong> vitro Transkripten.<br />
Die Ergebnisse s<strong>in</strong>d <strong>in</strong> der Abb. 5.49 gezeigt.<br />
120