Mechanistische Analysen zu Krankheits-korrelierten SNPs in ...
Mechanistische Analysen zu Krankheits-korrelierten SNPs in ...
Mechanistische Analysen zu Krankheits-korrelierten SNPs in ...
Erfolgreiche ePaper selbst erstellen
Machen Sie aus Ihren PDF Publikationen ein blätterbares Flipbook mit unserer einzigartigen Google optimierten e-Paper Software.
5 Ergebnisse<br />
kung der B<strong>in</strong>dung zwischen miRNA und Ziel-RNA. Diese Veränderungen können aber da<strong>zu</strong><br />
führen, dass bei gleicher Zugänglichkeit der miRNA B<strong>in</strong>destellen e<strong>in</strong> mismatch im seed<br />
Bereich ke<strong>in</strong>en messbaren E<strong>in</strong>fluss auf die B<strong>in</strong>dung <strong>zu</strong>r SNP-Variante hat. Dieser Zusammenhang<br />
stellt e<strong>in</strong>en möglichen Erklärungsansatz für die ähnliche pre-miR-155-vermittelte<br />
Repression beider kurzer AGTR1 Transkripte dar.<br />
Ergänzend <strong>zu</strong>r pre-miR-155 kamen zwei weitere miRNA-Tools mit bekannter Sequenz und<br />
ohne chemische Modifikationen <strong>zu</strong>r Anwendung (miR-155 und miR-155_SNP). Die Komplementaritäten<br />
deren guide Stränge mit den AGTR1-Varianten s<strong>in</strong>d der Abb. 5.2 dargestellt.<br />
Die miR-155 ist durch e<strong>in</strong>e Basenpaarung mit der AGTR1 A Variante an Position 5 ausgehend<br />
vom 5’-Ende der miRNA gekennzeichnet. Das AGTR1 C weist jedoch an dieser<br />
Position e<strong>in</strong>en C-U mismatch auf. Durch E<strong>in</strong>führung e<strong>in</strong>es U>G Basenaustausches <strong>in</strong>nerhalb<br />
des miR-155 guide Stranges an der Position 5 wird die miR-155_SNP generiert. Diese<br />
bildet e<strong>in</strong>en C-G match mit der AGTR1 C-Variante <strong>zu</strong>gleich aber e<strong>in</strong>en A-G mismatch mit<br />
der AGTR1 A-Variante.<br />
120<br />
110<br />
***<br />
*<br />
100<br />
***<br />
90<br />
83 80<br />
82<br />
85<br />
FL/RL <strong>in</strong> %<br />
90<br />
80<br />
76<br />
81<br />
77<br />
AGTR1 A<br />
AGTR1 C<br />
70<br />
60<br />
50<br />
kurz<br />
miR-155<br />
3′-UTR<br />
kurz<br />
miR-155_SNP<br />
3′-UTR<br />
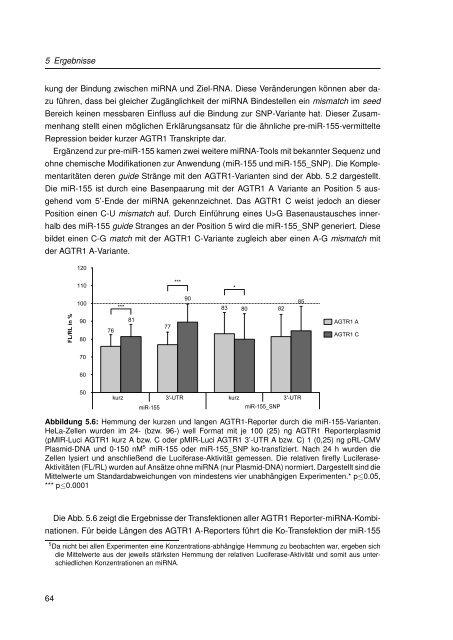
Abbildung 5.6: Hemmung der kurzen und langen AGTR1-Reporter durch die miR-155-Varianten.<br />
HeLa-Zellen wurden im 24- (bzw. 96-) well Format mit je 100 (25) ng AGTR1 Reporterplasmid<br />
(pMIR-Luci AGTR1 kurz A bzw. C oder pMIR-Luci AGTR1 3’-UTR A bzw. C) 1 (0,25) ng pRL-CMV<br />
Plasmid-DNA und 0-150 nM 5 miR-155 oder miR-155_SNP ko-transfiziert. Nach 24 h wurden die<br />
Zellen lysiert und anschließend die Luciferase-Aktivität gemessen. Die relativen firefly Luciferase-<br />
Aktivitäten (FL/RL) wurden auf Ansätze ohne miRNA (nur Plasmid-DNA) normiert. Dargestellt s<strong>in</strong>d die<br />
Mittelwerte um Standardabweichungen von m<strong>in</strong>destens vier unabhängigen Experimenten.* p≤0.05,<br />
*** p≤0.0001<br />
Die Abb. 5.6 zeigt die Ergebnisse der Transfektionen aller AGTR1 Reporter-miRNA-Komb<strong>in</strong>ationen.<br />
Für beide Längen des AGTR1 A-Reporters führt die Ko-Transfektion der miR-155<br />
5 Da nicht bei allen Experimenten e<strong>in</strong>e Konzentrations-abhängige Hemmung <strong>zu</strong> beobachten war, ergeben sich<br />
die Mittelwerte aus der jeweils stärksten Hemmung der relativen Luciferase-Aktivität und somit aus unterschiedlichen<br />
Konzentrationen an miRNA.<br />
64