Mechanistische Analysen zu Krankheits-korrelierten SNPs in ...
Mechanistische Analysen zu Krankheits-korrelierten SNPs in ...
Mechanistische Analysen zu Krankheits-korrelierten SNPs in ...
Erfolgreiche ePaper selbst erstellen
Machen Sie aus Ihren PDF Publikationen ein blätterbares Flipbook mit unserer einzigartigen Google optimierten e-Paper Software.
5.3 Untersuchungen im AGTR1-System<br />
5.3.7 RNA-Strukturprob<strong>in</strong>g<br />
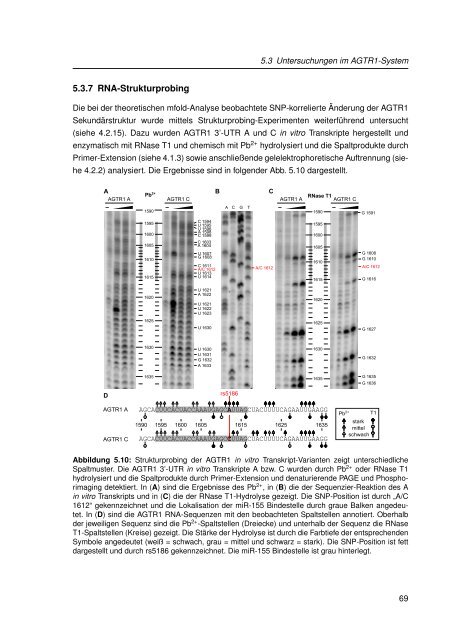
Die bei der theoretischen mfold-Analyse beobachtete SNP-korrelierte Änderung der AGTR1<br />
Sekundärstruktur wurde mittels Strukturprob<strong>in</strong>g-Experimenten weiterführend untersucht<br />
(siehe 4.2.15). Da<strong>zu</strong> wurden AGTR1 3’-UTR A und C <strong>in</strong> vitro Transkripte hergestellt und<br />
enzymatisch mit RNase T1 und chemisch mit Pb 2+ hydrolysiert und die Spaltprodukte durch<br />
Primer-Extension (siehe 4.1.3) sowie anschließende gelelektrophoretische Auftrennung (siehe<br />
4.2.2) analysiert. Die Ergebnisse s<strong>in</strong>d <strong>in</strong> folgender Abb. 5.10 dargestellt.<br />
A<br />
AGTR1 A<br />
-<br />
1590<br />
AGTR1 C<br />
-<br />
B<br />
A C G T<br />
C<br />
AGTR1 A<br />
-<br />
RNase T1<br />
AGTR1 C<br />
-<br />
1590<br />
G 1591<br />
1595<br />
1600<br />
1605<br />
1610<br />
1615<br />
C 1594<br />
U 1595<br />
U 1596<br />
A 1598<br />
C 1599<br />
C 1603<br />
A 1604<br />
U 1607<br />
G 1608<br />
C 1611<br />
A/C 1612<br />
U 1613<br />
U 1614<br />
A/C 1612<br />
1595<br />
1600<br />
1605<br />
1610<br />
1615<br />
G 1608<br />
G 1610<br />
A/C 1612<br />
G 1616<br />
1620<br />
Pb 2+ U 1623<br />
U 1621<br />
A 1622<br />
U 1621<br />
U 1622<br />
1620<br />
1625<br />
U 1630<br />
1625<br />
G 1627<br />
1630<br />
U 1630<br />
U 1631<br />
G 1632<br />
A 1633<br />
1630<br />
G 1632<br />
1635<br />
1635<br />
G 1635<br />
G 1636<br />
D<br />
rs5186<br />
AGTR1 A<br />
AGTR1 C<br />
AGCACUUCACUACCAAAUGAGCAUUAGCUACUUUUCAGAAUUGAAGG<br />
1590<br />
1595<br />
1600<br />
1605 1615 1625<br />
1635<br />
AGCACUUCACUACCAAAUGAGCCUUAGCUACUUUUCAGAAUUGAAGG<br />
Pb 2+<br />
stark<br />
mittel<br />
schwach<br />
T1<br />
Abbildung 5.10: Strukturprob<strong>in</strong>g der AGTR1 <strong>in</strong> vitro Transkript-Varianten zeigt unterschiedliche<br />
Spaltmuster. Die AGTR1 3’-UTR <strong>in</strong> vitro Transkripte A bzw. C wurden durch Pb 2+ oder RNase T1<br />
hydrolysiert und die Spaltprodukte durch Primer-Extension und denaturierende PAGE und Phosphorimag<strong>in</strong>g<br />
detektiert. In (A) s<strong>in</strong>d die Ergebnisse des Pb 2+ , <strong>in</strong> (B) die der Sequenzier-Reaktion des A<br />
<strong>in</strong> vitro Transkripts und <strong>in</strong> (C) die der RNase T1-Hydrolyse gezeigt. Die SNP-Position ist durch „A/C<br />
1612“ gekennzeichnet und die Lokalisation der miR-155 B<strong>in</strong>destelle durch graue Balken angedeutet.<br />
In (D) s<strong>in</strong>d die AGTR1 RNA-Sequenzen mit den beobachteten Spaltstellen annotiert. Oberhalb<br />
der jeweiligen Sequenz s<strong>in</strong>d die Pb 2+ -Spaltstellen (Dreiecke) und unterhalb der Sequenz die RNase<br />
T1-Spaltstellen (Kreise) gezeigt. Die Stärke der Hydrolyse ist durch die Farbtiefe der entsprechenden<br />
Symbole angedeutet (weiß = schwach, grau = mittel und schwarz = stark). Die SNP-Position ist fett<br />
dargestellt und durch rs5186 gekennzeichnet. Die miR-155 B<strong>in</strong>destelle ist grau h<strong>in</strong>terlegt.<br />
69