Mechanistische Analysen zu Krankheits-korrelierten SNPs in ...
Mechanistische Analysen zu Krankheits-korrelierten SNPs in ...
Mechanistische Analysen zu Krankheits-korrelierten SNPs in ...
Erfolgreiche ePaper selbst erstellen
Machen Sie aus Ihren PDF Publikationen ein blätterbares Flipbook mit unserer einzigartigen Google optimierten e-Paper Software.
5 Ergebnisse<br />
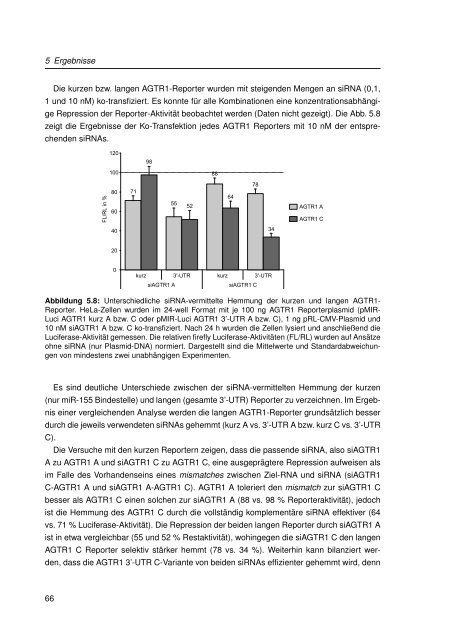
Die kurzen bzw. langen AGTR1-Reporter wurden mit steigenden Mengen an siRNA (0,1,<br />
1 und 10 nM) ko-transfiziert. Es konnte für alle Komb<strong>in</strong>ationen e<strong>in</strong>e konzentrationsabhängige<br />
Repression der Reporter-Aktivität beobachtet werden (Daten nicht gezeigt). Die Abb. 5.8<br />
zeigt die Ergebnisse der Ko-Transfektion jedes AGTR1 Reporters mit 10 nM der entsprechenden<br />
siRNAs.<br />
120<br />
98<br />
100<br />
88<br />
FL/RL <strong>in</strong> %<br />
80<br />
60<br />
71<br />
55<br />
52<br />
64<br />
78<br />
AGTR1 A<br />
AGTR1 C<br />
40<br />
34<br />
20<br />
0<br />
kurz 3′-UTR kurz 3′-UTR<br />
siAGTR1 A<br />
siAGTR1 C<br />
Abbildung 5.8: Unterschiedliche siRNA-vermittelte Hemmung der kurzen und langen AGTR1-<br />
Reporter. HeLa-Zellen wurden im 24-well Format mit je 100 ng AGTR1 Reporterplasmid (pMIR-<br />
Luci AGTR1 kurz A bzw. C oder pMIR-Luci AGTR1 3’-UTR A bzw. C), 1 ng pRL-CMV-Plasmid und<br />
10 nM siAGTR1 A bzw. C ko-transfiziert. Nach 24 h wurden die Zellen lysiert und anschließend die<br />
Luciferase-Aktivität gemessen. Die relativen firefly Luciferase-Aktivitäten (FL/RL) wurden auf Ansätze<br />
ohne siRNA (nur Plasmid-DNA) normiert. Dargestellt s<strong>in</strong>d die Mittelwerte und Standardabweichungen<br />
von m<strong>in</strong>destens zwei unabhängigen Experimenten.<br />
Es s<strong>in</strong>d deutliche Unterschiede zwischen der siRNA-vermittelten Hemmung der kurzen<br />
(nur miR-155 B<strong>in</strong>destelle) und langen (gesamte 3’-UTR) Reporter <strong>zu</strong> verzeichnen. Im Ergebnis<br />
e<strong>in</strong>er vergleichenden Analyse werden die langen AGTR1-Reporter grundsätzlich besser<br />
durch die jeweils verwendeten siRNAs gehemmt (kurz A vs. 3’-UTR A bzw. kurz C vs. 3’-UTR<br />
C).<br />
Die Versuche mit den kurzen Reportern zeigen, dass die passende siRNA, also siAGTR1<br />
A <strong>zu</strong> AGTR1 A und siAGTR1 C <strong>zu</strong> AGTR1 C, e<strong>in</strong>e ausgeprägtere Repression aufweisen als<br />
im Falle des Vorhandense<strong>in</strong>s e<strong>in</strong>es mismatches zwischen Ziel-RNA und siRNA (siAGTR1<br />
C-AGTR1 A und siAGTR1 A-AGTR1 C). AGTR1 A toleriert den mismatch <strong>zu</strong>r siAGTR1 C<br />
besser als AGTR1 C e<strong>in</strong>en solchen <strong>zu</strong>r siAGTR1 A (88 vs. 98 % Reporteraktivität), jedoch<br />
ist die Hemmung des AGTR1 C durch die vollständig komplementäre siRNA effektiver (64<br />
vs. 71 % Luciferase-Aktivität). Die Repression der beiden langen Reporter durch siAGTR1 A<br />
ist <strong>in</strong> etwa vergleichbar (55 und 52 % Restaktivität), woh<strong>in</strong>gegen die siAGTR1 C den langen<br />
AGTR1 C Reporter selektiv stärker hemmt (78 vs. 34 %). Weiterh<strong>in</strong> kann bilanziert werden,<br />
dass die AGTR1 3’-UTR C-Variante von beiden siRNAs effizienter gehemmt wird, denn<br />
66