Mechanistische Analysen zu Krankheits-korrelierten SNPs in ...
Mechanistische Analysen zu Krankheits-korrelierten SNPs in ...
Mechanistische Analysen zu Krankheits-korrelierten SNPs in ...
Sie wollen auch ein ePaper? Erhöhen Sie die Reichweite Ihrer Titel.
YUMPU macht aus Druck-PDFs automatisch weboptimierte ePaper, die Google liebt.
5 Ergebnisse<br />
A<br />
[ 32 P]-miR-224 guide<br />
[ 32 P]-miR-224_SNP guide<br />
TCF21 3'-UTR C<br />
TCF21 3'-UTR G<br />
-<br />
- TCF21 3'-UTR C TCF21 3'-UTR G<br />
0 6,66 20 40 120<br />
0 6,66 20 40 120<br />
0 80 t <strong>in</strong> m<strong>in</strong><br />
Komplex<br />
0 80 0 6,66 20 40 120 0 6,66 20 40 120<br />
miRNA<br />
B<br />
30<br />
% Komplex<br />
25<br />
20<br />
15<br />
10<br />
5<br />
0<br />
2,7 1,1<br />
24,2<br />
19,8<br />
20,9<br />
16,1<br />
0,40,6 0,6 1,2 0,4 1,3 0,5 1,6 0,5 2,4<br />
11,4<br />
8,4<br />
4,9<br />
6,5<br />
0 6,6 m<strong>in</strong> 20 m<strong>in</strong> 40 m<strong>in</strong> 80 m<strong>in</strong> 0 6,6 m<strong>in</strong> 20 m<strong>in</strong> 40 m<strong>in</strong> 80 m<strong>in</strong><br />
TCF21 3′-UTR C<br />
TCF21 3′-UTR G<br />
miR-224<br />
miR-224_SNP<br />
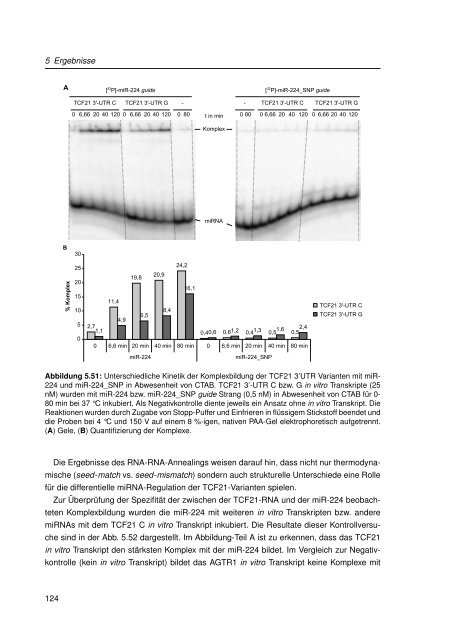
Abbildung 5.51: Unterschiedliche K<strong>in</strong>etik der Komplexbildung der TCF21 3’UTR Varianten mit miR-<br />
224 und miR-224_SNP <strong>in</strong> Abwesenheit von CTAB. TCF21 3’-UTR C bzw. G <strong>in</strong> vitro Transkripte (25<br />
nM) wurden mit miR-224 bzw. miR-224_SNP guide Strang (0,5 nM) <strong>in</strong> Abwesenheit von CTAB für 0-<br />
80 m<strong>in</strong> bei 37 °C <strong>in</strong>kubiert. Als Negativkontrolle diente jeweils e<strong>in</strong> Ansatz ohne <strong>in</strong> vitro Transkript. Die<br />
Reaktionen wurden durch Zugabe von Stopp-Puffer und E<strong>in</strong>frieren <strong>in</strong> flüssigem Stickstoff beendet und<br />
die Proben bei 4 °C und 150 V auf e<strong>in</strong>em 8 %-igen, nativen PAA-Gel elektrophoretisch aufgetrennt.<br />
(A) Gele, (B) Quantifizierung der Komplexe.<br />
Die Ergebnisse des RNA-RNA-Anneal<strong>in</strong>gs weisen darauf h<strong>in</strong>, dass nicht nur thermodynamische<br />
(seed-match vs. seed-mismatch) sondern auch strukturelle Unterschiede e<strong>in</strong>e Rolle<br />
für die differentielle miRNA-Regulation der TCF21-Varianten spielen.<br />
Zur Überprüfung der Spezifität der zwischen der TCF21-RNA und der miR-224 beobachteten<br />
Komplexbildung wurden die miR-224 mit weiteren <strong>in</strong> vitro Transkripten bzw. andere<br />
miRNAs mit dem TCF21 C <strong>in</strong> vitro Transkript <strong>in</strong>kubiert. Die Resultate dieser Kontrollversuche<br />
s<strong>in</strong>d <strong>in</strong> der Abb. 5.52 dargestellt. Im Abbildung-Teil A ist <strong>zu</strong> erkennen, dass das TCF21<br />
<strong>in</strong> vitro Transkript den stärksten Komplex mit der miR-224 bildet. Im Vergleich <strong>zu</strong>r Negativkontrolle<br />
(ke<strong>in</strong> <strong>in</strong> vitro Transkript) bildet das AGTR1 <strong>in</strong> vitro Transkript ke<strong>in</strong>e Komplexe mit<br />
124