Mechanistische Analysen zu Krankheits-korrelierten SNPs in ...
Mechanistische Analysen zu Krankheits-korrelierten SNPs in ...
Mechanistische Analysen zu Krankheits-korrelierten SNPs in ...
Sie wollen auch ein ePaper? Erhöhen Sie die Reichweite Ihrer Titel.
YUMPU macht aus Druck-PDFs automatisch weboptimierte ePaper, die Google liebt.
5.8 Untersuchungen im TCF21-System<br />
der jeweiligen Gleichung der Regressionsgeraden wurde die Menge an TCF21 C- bzw. G-<br />
Transkripten <strong>in</strong> den Proben berechnet. E<strong>in</strong>e Bee<strong>in</strong>flussung der Ergebnisse durch endogene<br />
TCF21-Transkripte konnte durch Messung der untransfizierten HeLa-Zellen ausgeschlossen<br />
werden (Daten nicht gezeigt). Zusätzlich wurde von jeder Probe die Expression des house<br />
keep<strong>in</strong>g Gens Glycer<strong>in</strong>aldehyd-3-phosphat-Dehydrogenase (GAPDH) gemessen.<br />
A<br />
ct-Werte<br />
10 2<br />
40<br />
C Primer<br />
10 3 10 4 10 5 10 6 10 7<br />
C Template<br />
35<br />
G Template<br />
30<br />
25 y = -1,9528Ln(x) + 48,985<br />
20<br />
15<br />
Kopien Template<br />
B<br />
ct-Werte<br />
10 2<br />
40<br />
G Primer<br />
10 3 10 4 10 5 10 6 10 7<br />
C Template<br />
35<br />
G Template<br />
30<br />
25<br />
y = -1,5893Ln(x) + 42,652<br />
20<br />
15<br />
Kopien Template<br />
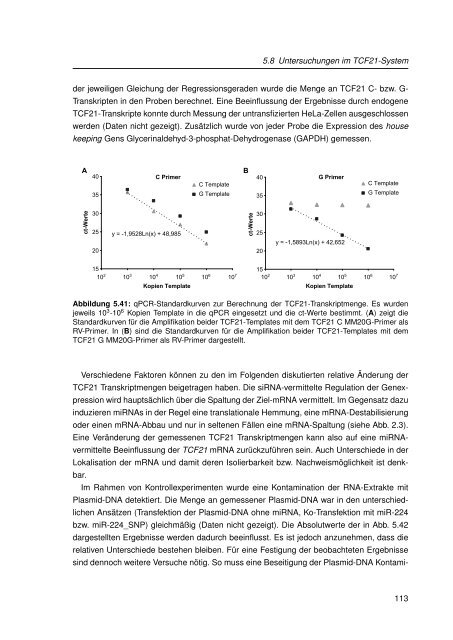
Abbildung 5.41: qPCR-Standardkurven <strong>zu</strong>r Berechnung der TCF21-Transkriptmenge. Es wurden<br />
jeweils 10 3 -10 6 Kopien Template <strong>in</strong> die qPCR e<strong>in</strong>gesetzt und die ct-Werte bestimmt. (A) zeigt die<br />
Standardkurven für die Amplifikation beider TCF21-Templates mit dem TCF21 C MM20G-Primer als<br />
RV-Primer. In (B) s<strong>in</strong>d die Standardkurven für die Amplifikation beider TCF21-Templates mit dem<br />
TCF21 G MM20G-Primer als RV-Primer dargestellt.<br />
Verschiedene Faktoren können <strong>zu</strong> den im Folgenden diskutierten relative Änderung der<br />
TCF21 Transkriptmengen beigetragen haben. Die siRNA-vermittelte Regulation der Genexpression<br />
wird hauptsächlich über die Spaltung der Ziel-mRNA vermittelt. Im Gegensatz da<strong>zu</strong><br />
<strong>in</strong>duzieren miRNAs <strong>in</strong> der Regel e<strong>in</strong>e translationale Hemmung, e<strong>in</strong>e mRNA-Destabilisierung<br />
oder e<strong>in</strong>en mRNA-Abbau und nur <strong>in</strong> seltenen Fällen e<strong>in</strong>e mRNA-Spaltung (siehe Abb. 2.3).<br />
E<strong>in</strong>e Veränderung der gemessenen TCF21 Transkriptmengen kann also auf e<strong>in</strong>e miRNAvermittelte<br />
Bee<strong>in</strong>flussung der TCF21 mRNA <strong>zu</strong>rück<strong>zu</strong>führen se<strong>in</strong>. Auch Unterschiede <strong>in</strong> der<br />
Lokalisation der mRNA und damit deren Isolierbarkeit bzw. Nachweismöglichkeit ist denkbar.<br />
Im Rahmen von Kontrollexperimenten wurde e<strong>in</strong>e Kontam<strong>in</strong>ation der RNA-Extrakte mit<br />
Plasmid-DNA detektiert. Die Menge an gemessener Plasmid-DNA war <strong>in</strong> den unterschiedlichen<br />
Ansätzen (Transfektion der Plasmid-DNA ohne miRNA, Ko-Transfektion mit miR-224<br />
bzw. miR-224_SNP) gleichmäßig (Daten nicht gezeigt). Die Absolutwerte der <strong>in</strong> Abb. 5.42<br />
dargestellten Ergebnisse werden dadurch bee<strong>in</strong>flusst. Es ist jedoch an<strong>zu</strong>nehmen, dass die<br />
relativen Unterschiede bestehen bleiben. Für e<strong>in</strong>e Festigung der beobachteten Ergebnisse<br />
s<strong>in</strong>d dennoch weitere Versuche nötig. So muss e<strong>in</strong>e Beseitigung der Plasmid-DNA Kontami-<br />
113