Mechanistische Analysen zu Krankheits-korrelierten SNPs in ...
Mechanistische Analysen zu Krankheits-korrelierten SNPs in ...
Mechanistische Analysen zu Krankheits-korrelierten SNPs in ...
Erfolgreiche ePaper selbst erstellen
Machen Sie aus Ihren PDF Publikationen ein blätterbares Flipbook mit unserer einzigartigen Google optimierten e-Paper Software.
4 Methoden<br />
Als Beschleuniger des RNA-RNA-Anneal<strong>in</strong>gs wurde das quaternäre Ammoniumsalz Cetyltrimethylammoniumbromid<br />
(CTAB) e<strong>in</strong>gesetzt. CTAB beschleunigt die K<strong>in</strong>etik von RNA-<br />
RNA-Interaktionen unter Bed<strong>in</strong>gungen, die weder die RNA-Struktur noch die sich ergebende<br />
Struktur-Funktions-Beziehung bee<strong>in</strong>flussen [127–130].<br />
5′<br />
t<br />
3′<br />
RNA-RNA Komplex<br />
3′<br />
5′<br />
3′<br />
5′<br />
ss miRNA bzw. siRNA<br />
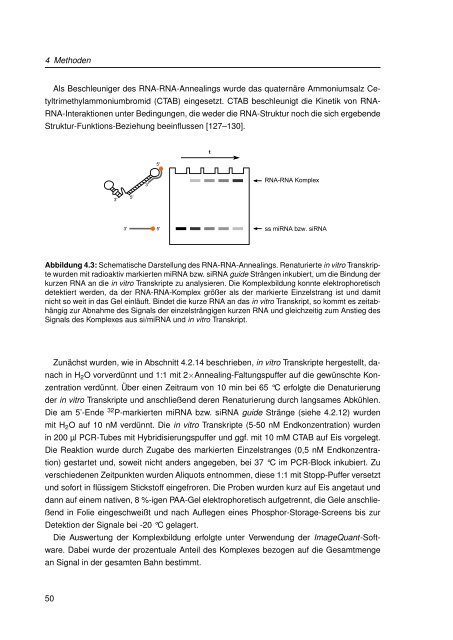
Abbildung 4.3: Schematische Darstellung des RNA-RNA-Anneal<strong>in</strong>gs. Renaturierte <strong>in</strong> vitro Transkripte<br />
wurden mit radioaktiv markierten miRNA bzw. siRNA guide Strängen <strong>in</strong>kubiert, um die B<strong>in</strong>dung der<br />
kurzen RNA an die <strong>in</strong> vitro Transkripte <strong>zu</strong> analysieren. Die Komplexbildung konnte elektrophoretisch<br />
detektiert werden, da der RNA-RNA-Komplex größer als der markierte E<strong>in</strong>zelstrang ist und damit<br />
nicht so weit <strong>in</strong> das Gel e<strong>in</strong>läuft. B<strong>in</strong>det die kurze RNA an das <strong>in</strong> vitro Transkript, so kommt es zeitabhängig<br />
<strong>zu</strong>r Abnahme des Signals der e<strong>in</strong>zelsträngigen kurzen RNA und gleichzeitig <strong>zu</strong>m Anstieg des<br />
Signals des Komplexes aus si/miRNA und <strong>in</strong> vitro Transkript.<br />
Zunächst wurden, wie <strong>in</strong> Abschnitt 4.2.14 beschrieben, <strong>in</strong> vitro Transkripte hergestellt, danach<br />
<strong>in</strong> H 2 O vorverdünnt und 1:1 mit 2×Anneal<strong>in</strong>g-Faltungspuffer auf die gewünschte Konzentration<br />
verdünnt. Über e<strong>in</strong>en Zeitraum von 10 m<strong>in</strong> bei 65 °C erfolgte die Denaturierung<br />
der <strong>in</strong> vitro Transkripte und anschließend deren Renaturierung durch langsames Abkühlen.<br />
Die am 5’-Ende 32 P-markierten miRNA bzw. siRNA guide Stränge (siehe 4.2.12) wurden<br />
mit H 2 O auf 10 nM verdünnt. Die <strong>in</strong> vitro Transkripte (5-50 nM Endkonzentration) wurden<br />
<strong>in</strong> 200 µl PCR-Tubes mit Hybridisierungspuffer und ggf. mit 10 mM CTAB auf Eis vorgelegt.<br />
Die Reaktion wurde durch Zugabe des markierten E<strong>in</strong>zelstranges (0,5 nM Endkonzentration)<br />
gestartet und, soweit nicht anders angegeben, bei 37 °C im PCR-Block <strong>in</strong>kubiert. Zu<br />
verschiedenen Zeitpunkten wurden Aliquots entnommen, diese 1:1 mit Stopp-Puffer versetzt<br />
und sofort <strong>in</strong> flüssigem Stickstoff e<strong>in</strong>gefroren. Die Proben wurden kurz auf Eis angetaut und<br />
dann auf e<strong>in</strong>em nativen, 8 %-igen PAA-Gel elektrophoretisch aufgetrennt, die Gele anschließend<br />
<strong>in</strong> Folie e<strong>in</strong>geschweißt und nach Auflegen e<strong>in</strong>es Phosphor-Storage-Screens bis <strong>zu</strong>r<br />
Detektion der Signale bei -20 °C gelagert.<br />
Die Auswertung der Komplexbildung erfolgte unter Verwendung der ImageQuant-Software.<br />
Dabei wurde der prozentuale Anteil des Komplexes bezogen auf die Gesamtmenge<br />
an Signal <strong>in</strong> der gesamten Bahn bestimmt.<br />
50