Mechanistische Analysen zu Krankheits-korrelierten SNPs in ...
Mechanistische Analysen zu Krankheits-korrelierten SNPs in ...
Mechanistische Analysen zu Krankheits-korrelierten SNPs in ...
Erfolgreiche ePaper selbst erstellen
Machen Sie aus Ihren PDF Publikationen ein blätterbares Flipbook mit unserer einzigartigen Google optimierten e-Paper Software.
5 Ergebnisse<br />
A<br />
firefly Luciferase „ESR1 kurz“<br />
ESR1 3′-UTR<br />
Pos. 71-101<br />
2330 2360<br />
5′ 3′<br />
BlpI<br />
AUG<br />
stop rs9341070<br />
1 653 2309 2355 2373<br />
SV40 Poly A<br />
MCS<br />
B<br />
5′-UTR<br />
CDS<br />
firefly Luciferase „ESR1 3′-UTR“<br />
3′-UTR<br />
ESR1 3′-UTR<br />
Pos. 1-1268<br />
2330<br />
3597<br />
5'<br />
3′<br />
AUG<br />
stop rs9341070<br />
1 653 2309<br />
2425<br />
3635<br />
5′-UTR CDS 3′-UTR<br />
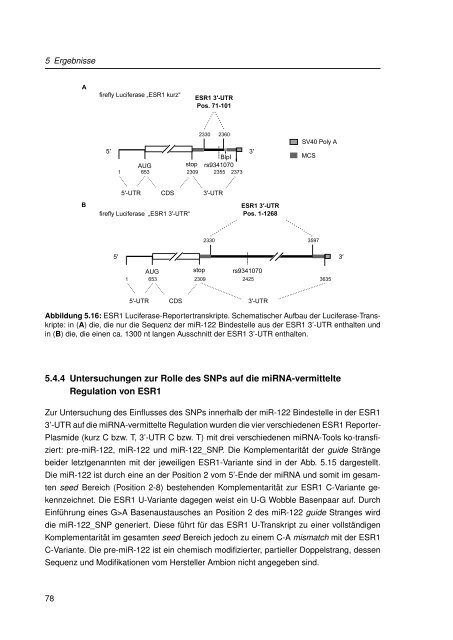
Abbildung 5.16: ESR1 Luciferase-Reportertranskripte. Schematischer Aufbau der Luciferase-Transkripte:<br />
<strong>in</strong> (A) die, die nur die Sequenz der miR-122 B<strong>in</strong>destelle aus der ESR1 3’-UTR enthalten und<br />
<strong>in</strong> (B) die, die e<strong>in</strong>en ca. 1300 nt langen Ausschnitt der ESR1 3’-UTR enthalten.<br />
5.4.4 Untersuchungen <strong>zu</strong>r Rolle des <strong>SNPs</strong> auf die miRNA-vermittelte<br />
Regulation von ESR1<br />
Zur Untersuchung des E<strong>in</strong>flusses des <strong>SNPs</strong> <strong>in</strong>nerhalb der miR-122 B<strong>in</strong>destelle <strong>in</strong> der ESR1<br />
3’-UTR auf die miRNA-vermittelte Regulation wurden die vier verschiedenen ESR1 Reporter-<br />
Plasmide (kurz C bzw. T, 3’-UTR C bzw. T) mit drei verschiedenen miRNA-Tools ko-transfiziert:<br />
pre-miR-122, miR-122 und miR-122_SNP. Die Komplementarität der guide Stränge<br />
beider letztgenannten mit der jeweiligen ESR1-Variante s<strong>in</strong>d <strong>in</strong> der Abb. 5.15 dargestellt.<br />
Die miR-122 ist durch e<strong>in</strong>e an der Position 2 vom 5’-Ende der miRNA und somit im gesamten<br />
seed Bereich (Position 2-8) bestehenden Komplementarität <strong>zu</strong>r ESR1 C-Variante gekennzeichnet.<br />
Die ESR1 U-Variante dagegen weist e<strong>in</strong> U-G Wobble Basenpaar auf. Durch<br />
E<strong>in</strong>führung e<strong>in</strong>es G>A Basenaustausches an Position 2 des miR-122 guide Stranges wird<br />
die miR-122_SNP generiert. Diese führt für das ESR1 U-Transkript <strong>zu</strong> e<strong>in</strong>er vollständigen<br />
Komplementarität im gesamten seed Bereich jedoch <strong>zu</strong> e<strong>in</strong>em C-A mismatch mit der ESR1<br />
C-Variante. Die pre-miR-122 ist e<strong>in</strong> chemisch modifizierter, partieller Doppelstrang, dessen<br />
Sequenz und Modifikationen vom Hersteller Ambion nicht angegeben s<strong>in</strong>d.<br />
78