Mechanistische Analysen zu Krankheits-korrelierten SNPs in ...
Mechanistische Analysen zu Krankheits-korrelierten SNPs in ...
Mechanistische Analysen zu Krankheits-korrelierten SNPs in ...
Erfolgreiche ePaper selbst erstellen
Machen Sie aus Ihren PDF Publikationen ein blätterbares Flipbook mit unserer einzigartigen Google optimierten e-Paper Software.
5 Ergebnisse<br />
[ 32 P]-siAGTR1 A guide<br />
[ 32 P]-siAGTR1 C guide<br />
Komplex<br />
unverdaut<br />
-<br />
-<br />
AGTR1 A<br />
10<br />
100<br />
- -<br />
-<br />
0.05<br />
-<br />
1<br />
-<br />
-<br />
AGTR1 C<br />
10<br />
100<br />
- -<br />
-<br />
0.05<br />
-<br />
1<br />
-<br />
- -<br />
- -<br />
passenger<br />
RNase T1 [u]<br />
RNase A [µg/ml]<br />
-<br />
-<br />
-<br />
passenger<br />
-<br />
-<br />
-<br />
-<br />
AGTR1 A<br />
10<br />
100<br />
- -<br />
-<br />
0.05<br />
-<br />
1<br />
-<br />
-<br />
AGTR1 C<br />
10<br />
100<br />
- -<br />
-<br />
0.05<br />
-<br />
1<br />
Komplex<br />
unverdaut<br />
Komplex<br />
T1 verdaut<br />
Komplex<br />
A verdaut<br />
ds siAGTR1<br />
siAGTR1<br />
guide<br />
Komplex<br />
T1 verdaut<br />
Komplex<br />
A verdaut<br />
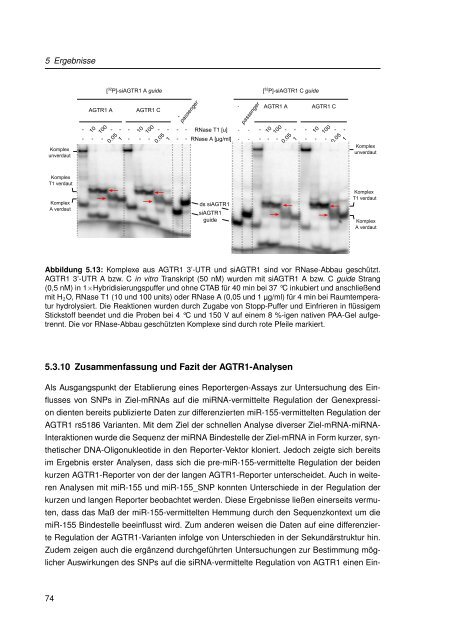
Abbildung 5.13: Komplexe aus AGTR1 3’-UTR und siAGTR1 s<strong>in</strong>d vor RNase-Abbau geschützt.<br />
AGTR1 3’-UTR A bzw. C <strong>in</strong> vitro Transkript (50 nM) wurden mit siAGTR1 A bzw. C guide Strang<br />
(0,5 nM) <strong>in</strong> 1×Hybridisierungspuffer und ohne CTAB für 40 m<strong>in</strong> bei 37 °C <strong>in</strong>kubiert und anschließend<br />
mit H 2 O, RNase T1 (10 und 100 units) oder RNase A (0,05 und 1 µg/ml) für 4 m<strong>in</strong> bei Raumtemperatur<br />
hydrolysiert. Die Reaktionen wurden durch Zugabe von Stopp-Puffer und E<strong>in</strong>frieren <strong>in</strong> flüssigem<br />
Stickstoff beendet und die Proben bei 4 °C und 150 V auf e<strong>in</strong>em 8 %-igen nativen PAA-Gel aufgetrennt.<br />
Die vor RNase-Abbau geschützten Komplexe s<strong>in</strong>d durch rote Pfeile markiert.<br />
5.3.10 Zusammenfassung und Fazit der AGTR1-<strong>Analysen</strong><br />
Als Ausgangspunkt der Etablierung e<strong>in</strong>es Reportergen-Assays <strong>zu</strong>r Untersuchung des E<strong>in</strong>flusses<br />
von <strong>SNPs</strong> <strong>in</strong> Ziel-mRNAs auf die miRNA-vermittelte Regulation der Genexpression<br />
dienten bereits publizierte Daten <strong>zu</strong>r differenzierten miR-155-vermittelten Regulation der<br />
AGTR1 rs5186 Varianten. Mit dem Ziel der schnellen Analyse diverser Ziel-mRNA-miRNA-<br />
Interaktionen wurde die Sequenz der miRNA B<strong>in</strong>destelle der Ziel-mRNA <strong>in</strong> Form kurzer, synthetischer<br />
DNA-Oligonukleotide <strong>in</strong> den Reporter-Vektor kloniert. Jedoch zeigte sich bereits<br />
im Ergebnis erster <strong>Analysen</strong>, dass sich die pre-miR-155-vermittelte Regulation der beiden<br />
kurzen AGTR1-Reporter von der der langen AGTR1-Reporter unterscheidet. Auch <strong>in</strong> weiteren<br />
<strong>Analysen</strong> mit miR-155 und miR-155_SNP konnten Unterschiede <strong>in</strong> der Regulation der<br />
kurzen und langen Reporter beobachtet werden. Diese Ergebnisse ließen e<strong>in</strong>erseits vermuten,<br />
dass das Maß der miR-155-vermittelten Hemmung durch den Sequenzkontext um die<br />
miR-155 B<strong>in</strong>destelle bee<strong>in</strong>flusst wird. Zum anderen weisen die Daten auf e<strong>in</strong>e differenzierte<br />
Regulation der AGTR1-Varianten <strong>in</strong>folge von Unterschieden <strong>in</strong> der Sekundärstruktur h<strong>in</strong>.<br />
Zudem zeigen auch die ergänzend durchgeführten Untersuchungen <strong>zu</strong>r Bestimmung möglicher<br />
Auswirkungen des <strong>SNPs</strong> auf die siRNA-vermittelte Regulation von AGTR1 e<strong>in</strong>en E<strong>in</strong>-<br />
74