Mechanistische Analysen zu Krankheits-korrelierten SNPs in ...
Mechanistische Analysen zu Krankheits-korrelierten SNPs in ...
Mechanistische Analysen zu Krankheits-korrelierten SNPs in ...
Sie wollen auch ein ePaper? Erhöhen Sie die Reichweite Ihrer Titel.
YUMPU macht aus Druck-PDFs automatisch weboptimierte ePaper, die Google liebt.
5 Ergebnisse<br />
A<br />
ESR1 C<br />
ESR1 U<br />
2200<br />
2200<br />
2225<br />
2325<br />
2250<br />
C<br />
2275<br />
2225<br />
2325<br />
2250<br />
U<br />
2275<br />
2300<br />
2300<br />
2175<br />
2175<br />
2150<br />
2150<br />
2350<br />
2350<br />
2125<br />
2125<br />
2400<br />
2375<br />
2400<br />
2375<br />
2100<br />
2100<br />
2425<br />
2425<br />
2075<br />
2075<br />
2450<br />
2450<br />
B<br />
G<br />
C<br />
G<br />
C<br />
2230<br />
C<br />
A<br />
A<br />
U<br />
G<br />
G<br />
A<br />
U<br />
2320<br />
U<br />
A<br />
U<br />
G<br />
U<br />
C<br />
U<br />
U<br />
C<br />
ESR1 C<br />
2240<br />
U<br />
A<br />
U<br />
C<br />
U<br />
2310<br />
C<br />
G<br />
C<br />
U<br />
G<br />
C<br />
A<br />
U<br />
C U<br />
A<br />
A<br />
C<br />
C<br />
U<br />
C<br />
GC<br />
2250<br />
G<br />
C<br />
G<br />
C<br />
U<br />
A<br />
U<br />
U<br />
2260<br />
C<br />
G<br />
G<br />
C<br />
G<br />
C<br />
2230<br />
G<br />
C<br />
2320<br />
U<br />
2240<br />
U<br />
A<br />
U<br />
A<br />
A<br />
G<br />
A<br />
C<br />
C<br />
G<br />
U<br />
U<br />
C<br />
U<br />
U<br />
U<br />
G<br />
U<br />
U<br />
C<br />
C<br />
ESR1 U<br />
U<br />
G<br />
C<br />
C<br />
A<br />
A<br />
C<br />
A<br />
C<br />
U<br />
U<br />
A<br />
2250<br />
U<br />
2310<br />
C<br />
G<br />
U<br />
C<br />
G<br />
C<br />
U<br />
G<br />
A<br />
U<br />
G<br />
C<br />
U<br />
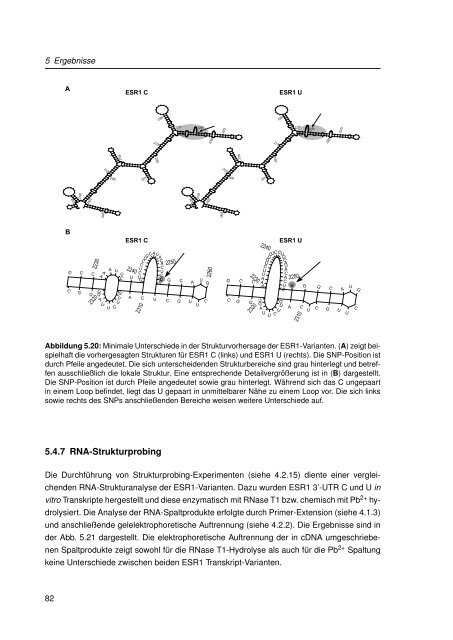
Abbildung 5.20: M<strong>in</strong>imale Unterschiede <strong>in</strong> der Strukturvorhersage der ESR1-Varianten. (A) zeigt beispielhaft<br />
die vorhergesagten Strukturen für ESR1 C (l<strong>in</strong>ks) und ESR1 U (rechts). Die SNP-Position ist<br />
durch Pfeile angedeutet. Die sich unterscheidenden Strukturbereiche s<strong>in</strong>d grau h<strong>in</strong>terlegt und betreffen<br />
ausschließlich die lokale Struktur. E<strong>in</strong>e entsprechende Detailvergrößerung ist <strong>in</strong> (B) dargestellt.<br />
Die SNP-Position ist durch Pfeile angedeutet sowie grau h<strong>in</strong>terlegt. Während sich das C ungepaart<br />
<strong>in</strong> e<strong>in</strong>em Loop bef<strong>in</strong>det, liegt das U gepaart <strong>in</strong> unmittelbarer Nähe <strong>zu</strong> e<strong>in</strong>em Loop vor. Die sich l<strong>in</strong>ks<br />
sowie rechts des <strong>SNPs</strong> anschließenden Bereiche weisen weitere Unterschiede auf.<br />
5.4.7 RNA-Strukturprob<strong>in</strong>g<br />
Die Durchführung von Strukturprob<strong>in</strong>g-Experimenten (siehe 4.2.15) diente e<strong>in</strong>er vergleichenden<br />
RNA-Strukturanalyse der ESR1-Varianten. Da<strong>zu</strong> wurden ESR1 3’-UTR C und U <strong>in</strong><br />
vitro Transkripte hergestellt und diese enzymatisch mit RNase T1 bzw. chemisch mit Pb 2+ hydrolysiert.<br />
Die Analyse der RNA-Spaltprodukte erfolgte durch Primer-Extension (siehe 4.1.3)<br />
und anschließende gelelektrophoretische Auftrennung (siehe 4.2.2). Die Ergebnisse s<strong>in</strong>d <strong>in</strong><br />
der Abb. 5.21 dargestellt. Die elektrophoretische Auftrennung der <strong>in</strong> cDNA umgeschriebenen<br />
Spaltprodukte zeigt sowohl für die RNase T1-Hydrolyse als auch für die Pb 2+ Spaltung<br />
ke<strong>in</strong>e Unterschiede zwischen beiden ESR1 Transkript-Varianten.<br />
82