Mechanistische Analysen zu Krankheits-korrelierten SNPs in ...
Mechanistische Analysen zu Krankheits-korrelierten SNPs in ...
Mechanistische Analysen zu Krankheits-korrelierten SNPs in ...
Erfolgreiche ePaper selbst erstellen
Machen Sie aus Ihren PDF Publikationen ein blätterbares Flipbook mit unserer einzigartigen Google optimierten e-Paper Software.
5.3 Untersuchungen im AGTR1-System<br />
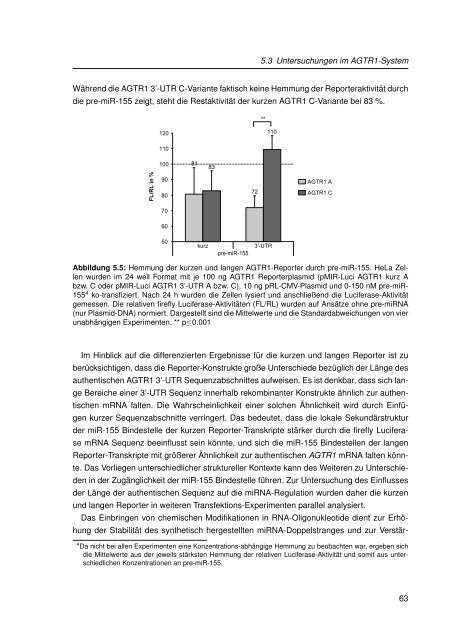
Während die AGTR1 3’-UTR C-Variante faktisch ke<strong>in</strong>e Hemmung der Reporteraktivität durch<br />
die pre-miR-155 zeigt, steht die Restaktivität der kurzen AGTR1 C-Variante bei 83 %.<br />
**<br />
120<br />
110<br />
110<br />
100<br />
81<br />
83<br />
FL/RL <strong>in</strong> %<br />
90<br />
80<br />
72<br />
AGTR1 A<br />
AGTR1 C<br />
70<br />
60<br />
50<br />
kurz<br />
pre-miR-155<br />
3′-UTR<br />
Abbildung 5.5: Hemmung der kurzen und langen AGTR1-Reporter durch pre-miR-155. HeLa Zellen<br />
wurden im 24 well Format mit je 100 ng AGTR1 Reporterplasmid (pMIR-Luci AGTR1 kurz A<br />
bzw. C oder pMIR-Luci AGTR1 3’-UTR A bzw. C), 10 ng pRL-CMV-Plasmid und 0-150 nM pre-miR-<br />
155 4 ko-transfiziert. Nach 24 h wurden die Zellen lysiert und anschließend die Luciferase-Aktivität<br />
gemessen. Die relativen firefly Luciferase-Aktivitäten (FL/RL) wurden auf Ansätze ohne pre-miRNA<br />
(nur Plasmid-DNA) normiert. Dargestellt s<strong>in</strong>d die Mittelwerte und die Standardabweichungen von vier<br />
unabhängigen Experimenten. ** p≤0.001<br />
Im H<strong>in</strong>blick auf die differenzierten Ergebnisse für die kurzen und langen Reporter ist <strong>zu</strong><br />
berücksichtigen, dass die Reporter-Konstrukte große Unterschiede bezüglich der Länge des<br />
authentischen AGTR1 3’-UTR Sequenzabschnittes aufweisen. Es ist denkbar, dass sich lange<br />
Bereiche e<strong>in</strong>er 3’-UTR Sequenz <strong>in</strong>nerhalb rekomb<strong>in</strong>anter Konstrukte ähnlich <strong>zu</strong>r authentischen<br />
mRNA falten. Die Wahrsche<strong>in</strong>lichkeit e<strong>in</strong>er solchen Ähnlichkeit wird durch E<strong>in</strong>fügen<br />
kurzer Sequenzabschnitte verr<strong>in</strong>gert. Das bedeutet, dass die lokale Sekundärstruktur<br />
der miR-155 B<strong>in</strong>destelle der kurzen Reporter-Transkripte stärker durch die firefly Luciferase<br />
mRNA Sequenz bee<strong>in</strong>flusst se<strong>in</strong> könnte, und sich die miR-155 B<strong>in</strong>destellen der langen<br />
Reporter-Transkripte mit größerer Ähnlichkeit <strong>zu</strong>r authentischen AGTR1 mRNA falten könnte.<br />
Das Vorliegen unterschiedlicher struktureller Kontexte kann des Weiteren <strong>zu</strong> Unterschieden<br />
<strong>in</strong> der Zugänglichkeit der miR-155 B<strong>in</strong>destelle führen. Zur Untersuchung des E<strong>in</strong>flusses<br />
der Länge der authentischen Sequenz auf die miRNA-Regulation wurden daher die kurzen<br />
und langen Reporter <strong>in</strong> weiteren Transfektions-Experimenten parallel analysiert.<br />
Das E<strong>in</strong>br<strong>in</strong>gen von chemischen Modifikationen <strong>in</strong> RNA-Oligonukleotide dient <strong>zu</strong>r Erhöhung<br />
der Stabilität des synthetisch hergestellten miRNA-Doppelstranges und <strong>zu</strong>r Verstär-<br />
4 Da nicht bei allen Experimenten e<strong>in</strong>e Konzentrations-abhängige Hemmung <strong>zu</strong> beobachten war, ergeben sich<br />
die Mittelwerte aus der jeweils stärksten Hemmung der relativen Luciferase-Aktivität und somit aus unterschiedlichen<br />
Konzentrationen an pre-miR-155.<br />
63