Mechanistische Analysen zu Krankheits-korrelierten SNPs in ...
Mechanistische Analysen zu Krankheits-korrelierten SNPs in ...
Mechanistische Analysen zu Krankheits-korrelierten SNPs in ...
Erfolgreiche ePaper selbst erstellen
Machen Sie aus Ihren PDF Publikationen ein blätterbares Flipbook mit unserer einzigartigen Google optimierten e-Paper Software.
5 Ergebnisse<br />
A<br />
[ 32 P]-miR-195 guide<br />
[ 32 P]-miR-135a guide<br />
MRAS 3'-UTR C<br />
MRAS 3'-UTR U<br />
-<br />
MRAS 3'-UTR C<br />
MRAS 3'-UTR U<br />
-<br />
0 10 30 60 120 0 10 30 60 120 0 120 t <strong>in</strong> s<br />
0 10 30 60 120 0 10 30 60 120 0 120<br />
Komplex<br />
miRNA<br />
B<br />
% Komplex<br />
12<br />
10<br />
8<br />
6<br />
4<br />
4,2<br />
3,2<br />
7,4<br />
5,0<br />
9,9<br />
7,6<br />
MRAS 3′-UTR C<br />
MRAS 3′-UTR U<br />
2<br />
0<br />
0,9<br />
1,6<br />
0,8 1,3<br />
0 10 s 30 s 60 s 120 s<br />
miR-195 guide<br />
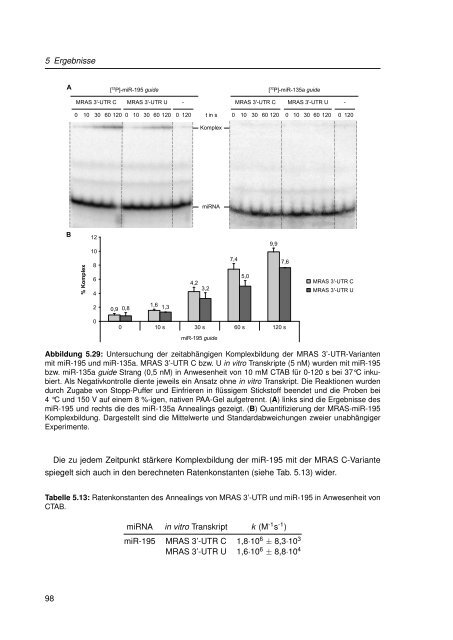
Abbildung 5.29: Untersuchung der zeitabhängigen Komplexbildung der MRAS 3’-UTR-Varianten<br />
mit miR-195 und miR-135a. MRAS 3’-UTR C bzw. U <strong>in</strong> vitro Transkripte (5 nM) wurden mit miR-195<br />
bzw. miR-135a guide Strang (0,5 nM) <strong>in</strong> Anwesenheit von 10 mM CTAB für 0-120 s bei 37°C <strong>in</strong>kubiert.<br />
Als Negativkontrolle diente jeweils e<strong>in</strong> Ansatz ohne <strong>in</strong> vitro Transkript. Die Reaktionen wurden<br />
durch Zugabe von Stopp-Puffer und E<strong>in</strong>frieren <strong>in</strong> flüssigem Stickstoff beendet und die Proben bei<br />
4 °C und 150 V auf e<strong>in</strong>em 8 %-igen, nativen PAA-Gel aufgetrennt. (A) l<strong>in</strong>ks s<strong>in</strong>d die Ergebnisse des<br />
miR-195 und rechts die des miR-135a Anneal<strong>in</strong>gs gezeigt. (B) Quantifizierung der MRAS-miR-195<br />
Komplexbildung. Dargestellt s<strong>in</strong>d die Mittelwerte und Standardabweichungen zweier unabhängiger<br />
Experimente.<br />
Die <strong>zu</strong> jedem Zeitpunkt stärkere Komplexbildung der miR-195 mit der MRAS C-Variante<br />
spiegelt sich auch <strong>in</strong> den berechneten Ratenkonstanten (siehe Tab. 5.13) wider.<br />
Tabelle 5.13: Ratenkonstanten des Anneal<strong>in</strong>gs von MRAS 3’-UTR und miR-195 <strong>in</strong> Anwesenheit von<br />
CTAB.<br />
miRNA <strong>in</strong> vitro Transkript k (M -1 s -1 )<br />
miR-195 MRAS 3’-UTR C 1,8·10 6 ± 8,3·10 3<br />
MRAS 3’-UTR U 1,6·10 6 ± 8,8·10 4<br />
98