Senza titolo-2 - aafvg associazione allevatori del friuli venezia giulia
Senza titolo-2 - aafvg associazione allevatori del friuli venezia giulia
Senza titolo-2 - aafvg associazione allevatori del friuli venezia giulia
Create successful ePaper yourself
Turn your PDF publications into a flip-book with our unique Google optimized e-Paper software.
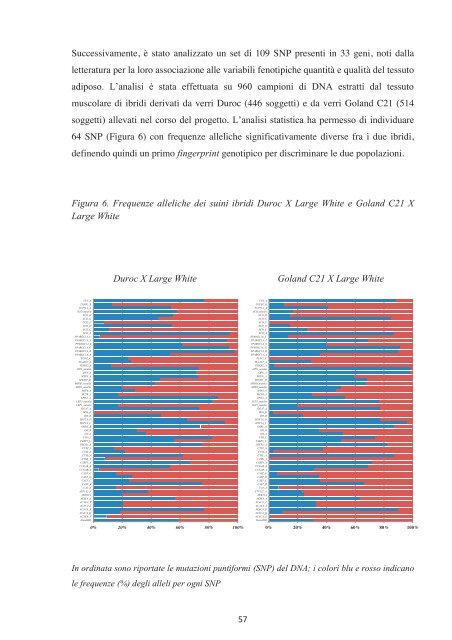
Successivamente, è stato analizzato un set di 109 SNP presenti in 33 geni, noti dalla<br />
letteratura per la loro <strong>associazione</strong> alle variabili fenotipiche quantità e qualità <strong>del</strong> tessuto<br />
adiposo. L’analisi è stata effettuata su 960 campioni di DNA estratti dal tessuto<br />
muscolare di ibridi derivati da verri Duroc (446 soggetti) e da verri Goland C21 (514<br />
soggetti) allevati nel corso <strong>del</strong> progetto. L’analisi statistica ha permesso di individuare<br />
64 SNP (Figura 6) con frequenze alleliche significativamente diverse fra i due ibridi,<br />
definendo quindi un primo fingerprint genotipico per discriminare le due popolazioni.<br />
Figura 6. Frequenze alleliche dei suini ibridi Duroc X Large White e Goland C21 X<br />
Large White<br />
Duroc X Large White Goland C21 X Large White<br />
TTN_A<br />
TGFB2_B<br />
TCF7L2_A<br />
SCD_natalia<br />
SCD_H<br />
SCD_G<br />
SCD_F<br />
SCD_D<br />
SCD_C<br />
SCD_A<br />
PPARGC1A_U<br />
PPARGC1A_T<br />
PPARGC1A_S<br />
PPARGC1A_G<br />
PPARGC1A_B<br />
PPARGC1A_A<br />
PLIN2_C<br />
PLA2G7_A<br />
PI3KR2_A<br />
OPN_natalia<br />
OPN_A<br />
MYPN_A<br />
MYOD1_D<br />
MYOD_natalia<br />
MYF6_natalia<br />
MYF6_A<br />
MC3R_A<br />
LPIN1_A<br />
LEP2_natalia<br />
LEP1_natalia<br />
ITGA7_A<br />
IRS4_B<br />
ID3_B<br />
HNF1A_D<br />
HNF1A_C<br />
GHRL_B<br />
GH_B<br />
GH_A<br />
FTO_L<br />
FABP3_U<br />
DECR1_A<br />
CTSZ_A<br />
CTSS_A<br />
CTSL_A<br />
CTSK_A<br />
CSRP3_A<br />
CCKAR_B<br />
CCKAR_A<br />
CAST_G<br />
CAST_D<br />
CAST_C<br />
CAST_B<br />
CA3_A<br />
ATP1A2_A<br />
AKR1C4<br />
ACSL4_A<br />
ACACA_T<br />
ACACA_S<br />
ACACA_R<br />
ACACA_Q<br />
ACACA_P<br />
3betaHSD<br />
0% 20% 40% 60% 80% 100%<br />
57<br />
TTN_A<br />
TGFB2_B<br />
TCF7L2_A<br />
SCD_natalia<br />
SCD_H<br />
SCD_G<br />
SCD_F<br />
SCD_D<br />
SCD_C<br />
SCD_A<br />
PPARGC1A_U<br />
PPARGC1A_T<br />
PPARGC1A_S<br />
PPARGC1A_G<br />
PPARGC1A_B<br />
PPARGC1A_A<br />
PLIN2_C<br />
PLA2G7_A<br />
PI3KR2_A<br />
OPN_natalia<br />
OPN_A<br />
MYPN_A<br />
MYOD1_D<br />
MYOD_natalia<br />
MYF6_natalia<br />
MYF6_A<br />
MC3R_A<br />
LPIN1_A<br />
LEP2_natalia<br />
LEP1_natalia<br />
ITGA7_A<br />
IRS4_B<br />
ID3_B<br />
HNF1A_D<br />
HNF1A_C<br />
GHRL_B<br />
GH_B<br />
GH_A<br />
FTO_L<br />
FABP3_U<br />
DECR1_A<br />
CTSZ_A<br />
CTSS_A<br />
CTSL_A<br />
CTSK_A<br />
CSRP3_A<br />
CCKAR_B<br />
CCKAR_A<br />
CAST_G<br />
CAST_D<br />
CAST_C<br />
CAST_B<br />
CA3_A<br />
ATP1A2_A<br />
AKR1C4<br />
ACSL4_A<br />
ACACA_T<br />
ACACA_S<br />
ACACA_R<br />
ACACA_Q<br />
ACACA_P<br />
3betaHSD<br />
0% 20% 40% 60% 80% 100%<br />
In ordinata sono riportate le mutazioni puntiformi (SNP) <strong>del</strong> DNA; i colori blu e rosso indicano<br />
le frequenze (%) degli alleli per ogni SNP