Dokument 2.pdf - OPUS-Datenbank - Universität Hohenheim
Dokument 2.pdf - OPUS-Datenbank - Universität Hohenheim
Dokument 2.pdf - OPUS-Datenbank - Universität Hohenheim
Sie wollen auch ein ePaper? Erhöhen Sie die Reichweite Ihrer Titel.
YUMPU macht aus Druck-PDFs automatisch weboptimierte ePaper, die Google liebt.
5.2 Untersuchungen zum Butanol-Metabolismus<br />
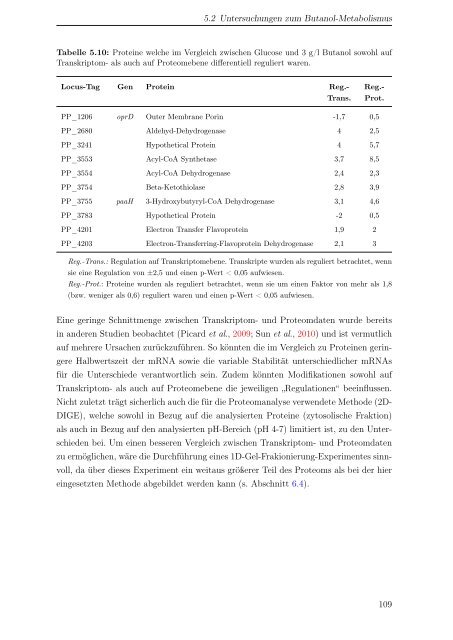
Tabelle 5.10: Proteine welche im Vergleich zwischen Glucose und 3 g/l Butanol sowohl auf<br />
Transkriptom- als auch auf Proteomebene differentiell reguliert waren.<br />
Trans.<br />
Locus-Tag Gen Protein Reg.-<br />
Reg.-<br />
Prot.<br />
PP_1206 oprD Outer Membrane Porin -1,7 0,5<br />
PP_2680 Aldehyd-Dehydrogenase 4 2,5<br />
PP_3241 Hypothetical Protein 4 5,7<br />
PP_3553 Acyl-CoA Synthetase 3,7 8,5<br />
PP_3554 Acyl-CoA Dehydrogenase 2,4 2,3<br />
PP_3754 Beta-Ketothiolase 2,8 3,9<br />
PP_3755 paaH 3-Hydroxybutyryl-CoA Dehydrogenase 3,1 4,6<br />
PP_3783 Hypothetical Protein -2 0,5<br />
PP_4201 Electron Transfer Flavoprotein 1,9 2<br />
PP_4203 Electron-Transferring-Flavoprotein Dehydrogenase 2,1 3<br />
Reg.-Trans.: Regulation auf Transkriptomebene. Transkripte wurden als reguliert betrachtet, wenn<br />
sie eine Regulation von ±2,5 und einen p-Wert < 0,05 aufwiesen.<br />
Reg.-Prot.: Proteine wurden als reguliert betrachtet, wenn sie um einen Faktor von mehr als 1,8<br />
(bzw. weniger als 0,6) reguliert waren und einen p-Wert < 0,05 aufwiesen.<br />
Eine geringe Schnittmenge zwischen Transkriptom- und Proteomdaten wurde bereits<br />
in anderen Studien beobachtet (Picard et al., 2009; Sun et al., 2010) und ist vermutlich<br />
auf mehrere Ursachen zurückzuführen. So könnten die im Vergleich zu Proteinen geringere<br />
Halbwertszeit der mRNA sowie die variable Stabilität unterschiedlicher mRNAs<br />
für die Unterschiede verantwortlich sein. Zudem könnten Modifikationen sowohl auf<br />
Transkriptom- als auch auf Proteomebene die jeweiligen „Regulationen“ beeinflussen.<br />
Nicht zuletzt trägt sicherlich auch die für die Proteomanalyse verwendete Methode (2D-<br />
DIGE), welche sowohl in Bezug auf die analysierten Proteine (zytosolische Fraktion)<br />
als auch in Bezug auf den analysierten pH-Bereich (pH 4-7) limitiert ist, zu den Unterschieden<br />
bei. Um einen besseren Vergleich zwischen Transkriptom- und Proteomdaten<br />
zu ermöglichen, wäre die Durchführung eines 1D-Gel-Frakionierung-Experimentes sinnvoll,<br />
da über dieses Experiment ein weitaus größerer Teil des Proteoms als bei der hier<br />
eingesetzten Methode abgebildet werden kann (s. Abschnitt 6.4).<br />
109